今年7月,被Meta解散的ESMFold团队成功另起炉灶,发布了他们最新的生命科学大模型ESM3,打出的slogan正是「用语言模型模拟5亿年进化。」
 图片
图片
论文地址:https://evolutionaryscale-public.s3.us-east-2.amazonaws.com/research/esm3.pdf
这个用途,很快就被生物学家们敏锐地捕捉到了。
最近发表的很多工作中,科学家们正在用AlphaFold和ESMFold等模型,重新绘制病毒谱系,探索到了一些令人惊讶的「亲缘关系」。
这些成果,不仅可以揭秘病毒家族的进化史,还能让我们更好地应对未来的生化风险。
 图片
图片
如果用传统方法,科学家们需要根据基因组比较的结果来理解病毒进化。
但是和哺乳动物比起来,病毒的进化速度可以说是快如闪电,尤其是基因由RNA组成的病毒,需要比对的基因组数量和复杂度就会急遽增加。
此外,病毒的进化不仅来源于基因突变,它们还可以从其他生物体获取遗传物质,这就辨识病毒「亲缘关系」的工作更加困难。看起来大不相同的基因序列,可能隐藏着病毒之间非常深层而遥远的关系。
相比病毒的基因,它们编码的蛋白质的形状或结构变化往往比较缓慢,然而英国格拉斯哥大学的分子病毒学家Joe Grove表示,在AlphaFold等工具出现之前,即使是整个病毒家族的蛋白质结构,也很难靠传统方法完成研究和比较。
Grove和他的团队最近在Nature上发表的一篇论文,正是借助了大模型的力量,通过糖蛋白的结构揭示了黄病毒科的进化史。
 图片
图片
论文地址:https://www.nature.com/articles/s41586-024-07899-8
黄病毒包括丙型肝炎病毒、登革热病毒和寨卡病毒,以及一些主要的动物病原体,还有一些可能对人类健康构成新威胁的物种。
病毒如何进入细胞
自从疫苗大范围接种后,丙肝成为了我们不太熟悉的一种传染病,但这个病毒每年依旧造成了数十万人的死亡。
如果要开发更为有效的丙肝疫苗,我们就需要理解黄病毒是凭借哪个蛋白质进入细胞的(其中就包含糖蛋白),这些蛋白质也同样决定了病毒能够感染哪些宿主。
如果仅在序列层面研究、比对,你会发现各个病毒的蛋白质差异如此之大,很难找到有意义的联系。但如果借助生物大模型的蛋白质结构预测功能,这个难题将迎刃而解。
研究人员使用DeepMind的AlphaFold 2模型,和Meta开发的结构预测工具ESMFold,为458种黄病毒的蛋白质生成了超过3.3万个预测结构。
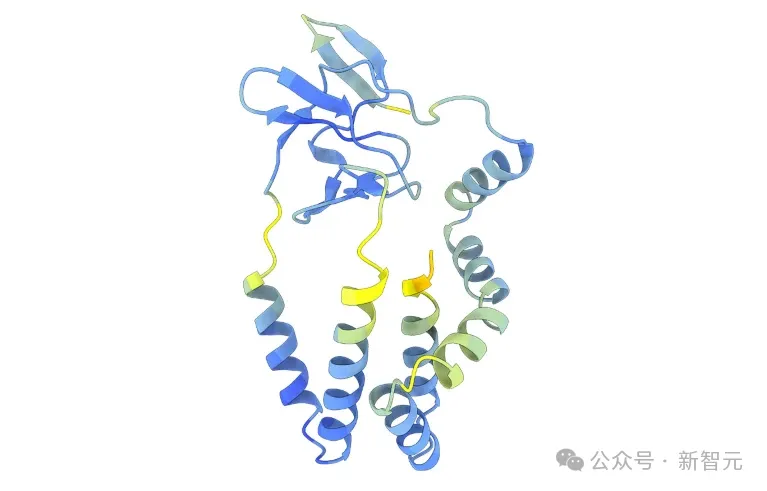 丙型肝炎病毒糖蛋白结构预测
丙型肝炎病毒糖蛋白结构预测
之所以同时使用AlphaFold和ESMFold两种模型,是由于二者之间的一个本质差异。
AlphaFold的输入需要依赖于相似蛋白质的多个序列,但ESMFold不同,它是在数千万个蛋白质序列上训练的「蛋白质语言模型」,可以只接受耽搁序列作为输入,因而非常适合深入分析那些最「神秘」的病毒。
这些结构的预测结果让研究人员们发现了一些意想不到的联系,有些和黄病毒看似八竿子打不着的亲戚,也能用类似蛋白作为「钥匙」来进入细胞。
比如,丙肝使用的细胞感染系统和瘟病毒就非常类似,包括比较经典的猪瘟,以及其他的动物病原体。
AI辅助工具还能告诉我们,丙肝和瘟病毒所用的「进入系统」和其他病毒非常不同。对此,Grove也很难做出解释:「对于丙型肝炎和它的亲戚们,我们不知道它们的进入系统来自哪里,可能是那些病毒很久以前发明的。」
从细菌中得到「盗版」蛋白
除了瘟病毒,预测的结构还帮黄病毒找到了两个「亲戚」——寨卡病毒和登革热病毒,它们的进入蛋白似乎有相同的起源;此外,黄病毒似乎还从细菌那里「窃取」了一种酶并据为己有。
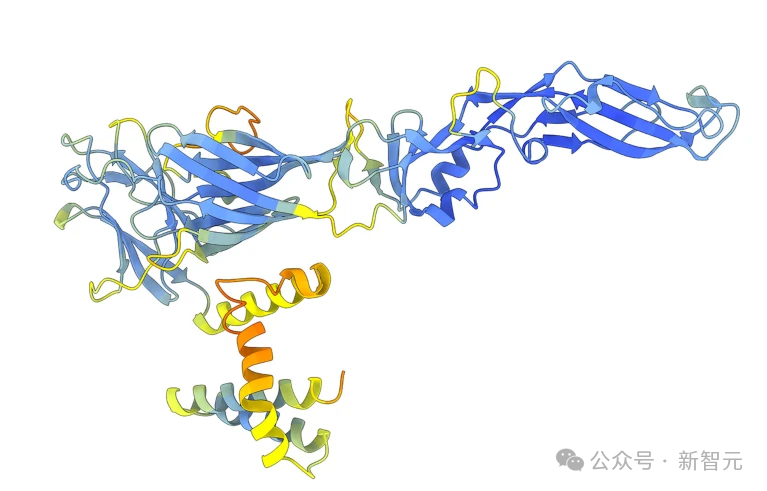 使用ColabFold–AlpahFold2预测登革热病毒蛋白的结构
使用ColabFold–AlpahFold2预测登革热病毒蛋白的结构
此前,悉尼大学病毒学家Mary Petrone的团队也曾在一种奇怪的黄病毒中发现了类似的「盗窃」行为。
她表示,「在黄病毒的进化过程中,『基因盗取』可能发挥了比我们之前认为的更大的塑造作用。」
瑞士洛桑大学的计算生物学家David Moi还指出,考虑到AI辅助工具未被发掘的潜力,黄病毒研究只是冰山一角。
借助人工智能,其他病毒,甚至很多细胞生物的进化历史都很可能被重写。
「我们将用新一代工具重新讲述它们的故事。既然我们现在能看得更远,所有这些生物的进化历史都需要更新。」
在生命科学的很多未解之谜中,AI所迸发出的巨大能量让我们看到了答案的曙光,也让我们期待着故事被改写的那一天。

























