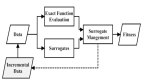
在构建机器学习模型时,特征选择是一个关键的预处理步骤。使用全部特征往往会导致过拟合、增加计算复杂度等问题。因此,我们需要从原始特征集中选择一个最优子集,以提高模型的泛化性能和效率。
特征选择的目标是找到一个二元掩码向量,对应每个特征的保留(1)或剔除(0)。例如,对于10个特征,这个掩码向量可能是[1,0,1,1,0,0,1,0,1,0]。我们需要通过某种优化方法,寻找一个使目标函数(如模型的贝叶斯信息准则BIC)最小化的最优掩码。
当特征数量较小时,我们可以使用暴力搜索等枚举方法。但随着特征数量的增加,搜索空间将呈指数级增长,枚举搜索将变得无法承受。这时我们需要借助启发式优化算法,例如遗传算法、模拟退火等,在有限时间内找到一个近似最优解。
本文以Kaggle房价数据集(213个特征)为例,比较了经典的序贯特征选择算法(SFS)和一种称为进化策略(CMA-ES)的启发式优化算法在特征选择任务上的表现。实验结果表明,CMA-ES相比SFS能更快地converge到更优的特征子集,从而获得更小的BIC值和更佳的模型泛化性能
GA - 遗传算法
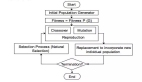
遗传算法是一种启发式优化算法,其灵感源自达尔文的进化论和自然选择理论。在自然界中,生物个体通过基因遗传,携带有利于生存和繁衍的特征,经过漫长的自然选择过程,适者生存,不断进化。类似地,遗传算法也是通过模拟这一自然进化过程,对问题的潜在解决方案(个体)进行遗传、变异和选择,最终得到满足目标条件的最优或近似最优解。
以特征选择为例,假设我们有N个特征,需要确定一个长度为N的二进制向量[1, 0, 0, 1, 1, 1, ...]来表示选择(1)或剔除(0)每个特征,目标是找到一个使成本函数或目标函数最小化的最优特征组合。我们可以将每个这样的二进制向量看作一个"个体",每个位置上的0或1就是该个体的一个"基因"。
遗传算法基本步骤:
- 评估:计算每个个体的适应度(目标函数值)。
- 选择:根据适应度挑选出若干个体作为父代。
- 交叉:随机选择两个父代个体,按一定交叉方式组合它们的基因,产生新的个体(子代)。
- 变异:以一定的小概率改变子代个体中的某些基因值。
- 替换:用新产生的子代个体替换种群中适应度较低的个体。
通过不断迭代上述过程,种群中个体的平均适应度就会不断提高,最终可以得到满足要求的最优或近似最优解。
一些关键概念和组件:
- 个体(Individual):表示种群中的一个候选解,通常用染色体(一串数字或字符)来表示。
- 种群(Population):包含多个个体的集合。
- 适应度函数(Fitness Function):用于评估每个个体的优劣程度。
- 选择(Selection):根据适应度函数的结果,从当前种群中选择部分个体作为父代,用于产生下一代种群。
- 交叉(Crossover):将两个父代个体的染色体进行组合,产生一个或多个新的子代个体。
- 变异(Mutation):通过改变个体染色体上的某些位,增加种群的多样性。
- 最佳个体(Hall of Fame):用于保存迄今为止找到的最佳个体,防止在进化过程中被淘汰或丢失。
- 优秀个体(Elitism):在每一代中,直接将当前种群中的优秀个体传递到下一代,而不需要经过选择、交叉和变异等操作。
遗传算法首先随机生成一组这样的个体,构成初始种群。比如,如果N=12,种群数量为8,那么初始种群可能包含8个长度为12的随机二进制向量。
 遗传算法-种群
遗传算法-种群
群体创建后,通过目标函数对每个个体进行评估。
接下来是选择阶段,旨在保留具有良好特征的个体,淘汰表现不佳的个体。这一过程有多种策略可供选择,从简单的排序法到更加复杂的随机锦标赛选择等。选择策略需要权衡探索(尝试新的解决方案)和利用(保留已知的良好解决方案)之间的平衡。遗传算法的核心在于通过有效探索来寻找最优解。常见的选择技术包括:锦标赛选择、轮盘赌选择、等级选择等。
经过选择后,遗传算法会在保留的优秀个体基因库中引入变异,以产生新的候选解。主要有两种变异技术:交叉和突变。
交叉是模仿生物交配过程,将两个亲代个体的部分基因随机组合,产生新的子代个体。这一过程能够将亲代的良好特征组合在一起,有望产生更优秀的后代。
突变则是对个体的基因做出少量随机改变,以引入新的多样性,避免群体过早收敛于局部最优解。
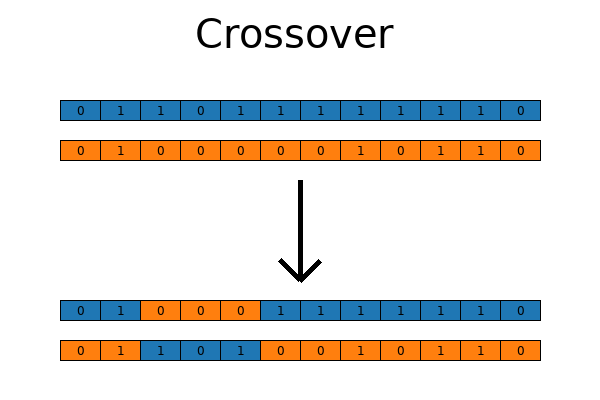 遗传算法-交叉
遗传算法-交叉
变异,同样是指自然界中遗传物质发生随机突变,基因库中引入新的价值,从而增加其多样性。
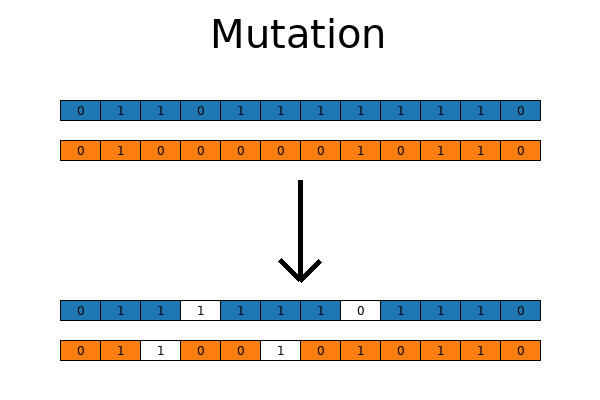 遗传算法-变异
遗传算法-变异
之后,算法循环往复:再次通过目标函数评估个体,进行选择,然后是交叉、变异等。
可以使用各种停止标准:如果目标函数在一定代数内停止改进,循环就会中断。或者你也可以设定一个硬性的停止条件,即已评估的总代数。或者以时间为基础,或者观察外部信号等。无论如何,具有最佳目标值的个体都应被视为问题的解决方案。
在遗传算法(GA)中,随机选择技术有时可能会由于偶然因素而放弃当代种群中最优秀的个体。尽管发生概率不高,但这种情况确实存在。这就是引入"精英主义"策略的目的,它确保无论如何,种群中的优秀个体都会被保留下来。然而,过度使用精英主义也可能导致算法陷入局部最优解,从而错失全局最优解。GA本质上是一种探索性算法,根据我有限的经验,为其引入过多偏见并不利于寻找最优解。GA本身就提供了大量的算法变体可供尝试和探索。
在GA中,我们可以调整以下几个超参数:
- 种群规模(个体数量)
- 基因突变概率
- 个体交叉概率
- 个体选择策略等
手动尝试不同超参数组合是寻找最优参数的一种方式。另一种高级方法是将GA封装到优化框架(如Optuna)中,利用框架自动搜索最佳超参数,但这种方式计算代价较高。
无论采用何种方式,GA都为我们提供了一种灵活有趣的优化探索方式,让我们可以充分发挥创意。
特征选择的GA代码
下面是一个可用于特征选择的简单 GA 代码。它使用了功能强大的 deap 库,但学习曲线可能比较陡峭。不过,这个简单的版本应该足够清晰。
上下滑动查看更多源码# to maximize the objective
# fitness_weights = 1.0
# to minimize the objective
fitness_weights = -1.0
# copy the original dataframes into local copies, once
X_ga = X.copy()
y_ga = y.copy()
# 'const' (the first column) is not an actual feature, do not include it
X_features = X_ga.columns.to_list()[1:]
try:
del creator.FitnessMax
del creator.Individual
except Exception as e:
pass
creator.create("FitnessMax", base.Fitness, weights=(fitness_weights,))
creator.create(
"Individual", array.array, typecode='b', fitness=creator.FitnessMax
)
try:
del toolbox
except Exception as e:
pass
toolbox = base.Toolbox()
# Attribute generator
toolbox.register("attr_bool", random.randint, 0, 1)
# Structure initializers
toolbox.register(
"individual",
tools.initRepeat,
creator.Individual,
toolbox.attr_bool,
len(X_features),
)
toolbox.register("population", tools.initRepeat, list, toolbox.individual)
def evalOneMax(individual):
# objective function
# create True/False selector list for features
# and add True at the start for 'const'
cols_select = [True] + [i == 1 for i in list(individual)]
# fit model using the features selected from the individual
lin_mod = sm.OLS(y_ga, X_ga.loc[:, cols_select], hascnotallow=True).fit()
return (lin_mod.bic,)
toolbox.register("evaluate", evalOneMax)
toolbox.register("mate", tools.cxTwoPoint)
toolbox.register("mutate", tools.mutFlipBit, indpb=0.05)
toolbox.register("select", tools.selTournament, tournsize=3)
random.seed(0)
pop = toolbox.population(n=300)
hof = tools.HallOfFame(1)
pop, log = algorithms.eaSimple(
pop, toolbox, cxpb=0.5, mutpb=0.2, ngen=10, halloffame=hof, verbose=True
)
best_individual_ga_small = list(hof[0])
best_features_ga_small = [
X_features[i] for i, val in enumerate(best_individual_ga_small) if val == 1
]
best_objective_ga_small = (
sm.OLS(y_ga, X_ga[['const'] + best_features_ga_small], hascnotallow=True)
.fit()
.bic
)
print(f'best objective: {best_objective_ga_small}')
print(f'best features: {best_features_ga_small}')代码使用对象来定义个体和整个种群,并采取评估(目标函数)、交叉/交配、突变和选择的策略。起初,代码从包含 300 个个体的种群开始,然后调用 eaSimple() 函数(该函数包含了交叉、变异和选择的预设序列),并且仅运行 10 代以简化过程。名人堂的规模为 1,它会保留绝对最优秀的个体,以防它们在选择等过程中被意外变异或跳过。
名人堂并非精英主义,而是从群体中复制最优秀的个体,并仅在锡罐中保留一个非活动副本。精英主义则是将活跃种群中最优秀的个体一代一代地保留下来。
这个简单的代码很容易理解,但效率很低。下面有更复杂版本的 GA 代码,更复杂、更优化的代码 1000 代后。
# to maximize the objective
# fitness_weights = 1.0
# to minimize the objective
fitness_weights = -1.0
# copy the original dataframes into local copies, once
X_ga = X.astype(float).copy()
y_ga = y.astype(float).copy()
# 'const' (the first column) is not an actual feature, do not include it
X_features = X_ga.columns.to_list()[1:]
try:
del creator.FitnessMax
del creator.Individual
except Exception as e:
pass
creator.create("FitnessMax", base.Fitness, weights=(fitness_weights,))
creator.create(
"Individual", array.array, typecode='b', fitness=creator.FitnessMax
)
try:
del toolbox
except Exception as e:
pass
toolbox = base.Toolbox()
# Attribute generator
toolbox.register("attr_bool", random.randint, 0, 1)
# Structure initializers
toolbox.register(
"individual",
tools.initRepeat,
creator.Individual,
toolbox.attr_bool,
len(X_features),
)
toolbox.register("population", tools.initRepeat, list, toolbox.individual)
def evalMany(individuals):
# individuals is an array of shape (n_individuals, n_features)
# transform it into a list of lists:
# - a list of individuals
# - each individual is a list of feature selectors
ind_list = [list(i) for i in list(individuals)]
ret = []
for ind in ind_list:
# create a list of True/False feature selectors from each individual
# pre-pend True to always select the 'const' feature
cols_select = [True] + [i == 1 for i in list(ind)]
# fit model using the features selected from the individual
lin_mod = sm.OLS(y_ga, X_ga.loc[:, cols_select], hascnotallow=True).fit()
ret.append((lin_mod.bic,))
return ret
# multiprocess pool to evaluate individuals
def joblib_map(f, njobs, *iters):
return Parallel(n_jobs=njobs)(delayed(f)(*args) for args in zip(*iters))
def selElitistAndTournament(
individuals, k_tournament, k_elitist=0, tournsize=3
):
# elitist tournament
# in addition to the regular tournament, ensure the top #k_elistist individuals are preserved
return tools.selBest(individuals, k_elitist) + tools.selTournament(
individuals, k_tournament, tournsize
)
# Hyperparameters
population_size = 1000
crossover_probability = 0.5
individual_mutation_probability = 0.2
gene_mutation_probability = 0.1
tournament_size = 3
elite_size = 0
n_jobs = os.cpu_count()
# register the pool as the mapper
# and the custom function as the evaluator
toolbox.register("map_multi", joblib_map)
toolbox.register("evaluate", evalMany)
toolbox.register("mate", tools.cxTwoPoint)
toolbox.register("mutate", tools.mutFlipBit, indpb=gene_mutation_probability)
# toolbox.register("select", tools.selTournament, tournsize=tournament_size)
# selection with tournament and optional elitism
toolbox.register(
"select",
selElitistAndTournament,
k_tournament=population_size - elite_size,
k_elitist=elite_size,
tournsize=tournament_size,
)
random.seed(0)
population = toolbox.population(n=population_size)
hall_of_fame = tools.HallOfFame(1)
objective_runs_ga = 0
# Evaluate the individuals with an invalid fitness
invalid_ind = [ind for ind in population if not ind.fitness.valid]
# split the population in a list with n_jobs elements
# each element is an array containing multiple individuals
# send individuals to the evaluator
fitnesses_nested = toolbox.map_multi(
toolbox.evaluate, n_jobs, np.array_split(invalid_ind, n_jobs)
)
objective_runs_ga += len(invalid_ind)
fitnesses = []
for l in fitnesses_nested:
fitnesses += l
for ind, fit in zip(invalid_ind, fitnesses):
ind.fitness.values = fit
hall_of_fame.update(population)
n_gen = 1000
best_objective_per_gen_ga = np.full(n_gen, np.nan)
best_objective_ga = np.nan
best_generation_ga = 0
gene_values_mean = np.zeros((n_gen, len(X_features)))
gene_maes = np.full(n_gen, np.nan)
time_to_best_ga = np.inf
# Begin the generational process
iterator = tqdm(range(1, n_gen + 1), desc='generation')
t_start = time.time()
for gen in iterator:
t_start_loop = time.time()
# Select the next generation of individuals
offspring = toolbox.select(population)
# Vary the pool of individuals via cross-over and mutation
offspring = algorithms.varAnd(
offspring,
toolbox,
crossover_probability,
individual_mutation_probability,
)
# Evaluate the individuals with an invalid fitness
invalid_ind = [ind for ind in offspring if not ind.fitness.valid]
# split the population in a list with n_jobs elements
# each element is an array containing multiple individuals
# send list to the evaluator pool
fitnesses_nested = toolbox.map_multi(
toolbox.evaluate, n_jobs, np.array_split(invalid_ind, n_jobs)
)
objective_runs_ga += len(invalid_ind)
fitnesses = []
for l in fitnesses_nested:
fitnesses += l
for ind, fit in zip(invalid_ind, fitnesses):
ind.fitness.values = fit
# Update the hall of fame with the generated individuals
hall_of_fame.update(offspring)
# Replace the current population by the offspring
population[:] = offspring
t_end_loop = time.time()
# record the mean gene values across the population
gene_values_mean[gen - 1, :] = np.array(population).mean(axis=0)
if gen >= 2:
gene_maes[gen - 1] = mean_absolute_error(
gene_values_mean[gen - 2, :], gene_values_mean[gen - 1, :]
)
# pick best individual for stats recording
best_individual_ga = tools.selBest(population, 1)[0]
best_objective_per_gen_ga[gen - 1] = best_individual_ga.fitness.values[0]
if (
best_objective_ga is np.nan
or fitness_weights * best_individual_ga.fitness.values[0]
> fitness_weights * best_objective_ga
):
best_objective_ga = best_individual_ga.fitness.values[0]
best_generation_ga = gen
time_to_best_ga = t_end_loop - t_start
print(
f'gen: {gen:4n}, curr/prev gene MAE: {gene_maes[gen - 1]:.4f}, new best objective: {best_objective_ga:.4f}, time to best: {time_to_best_ga:.4f}'
)
if os.path.isfile('break'):
# to gracefully break the loop, manually create a file called 'break'
print(f'Found break file, stopping now.')
iterator.close()
break
g_completed_ga = gen
best_individual_ga = list(hall_of_fame[0])
best_features_ga = [
X_features[i] for i, val in enumerate(best_individual_ga) if val == 1
]
best_objective_ga = (
sm.OLS(y_ga, X_ga[['const'] + best_features_ga], hascnotallow=True).fit().bic
)
## 强烈建议关注#公众号:数据STUDIO 更多优质内容每日更新!best objective: 33705.569572544795
best generation: 787
objective runs: 600525
time to best: 158.027 sec同样,在选择任何特征之前的基准 BIC 计算:
上下滑动查看更多源码print(f'best objective: {best_objective_ga}')
print(f'best generation: {best_generation_ga}')
print(f'objective runs: {objective_runs_ga}')
print(f'time to best: {time_to_best_ga:.3f} sec')
print(f'best features: {best_features_ga}')
gvm_df = (
pd.DataFrame(
gene_values_mean,
columns=X_features,
index=range(1, gene_values_mean.shape[0] + 1),
)
.sort_index(axis=1)
.iloc[:g_completed_ga]
)
if g_completed_ga > 20:
x_ticks = list(range(1, g_completed_ga + 1, round(g_completed_ga / 20)))
else:
x_ticks = list(range(1, g_completed_ga + 1))
if x_ticks[-1] != g_completed_ga:
x_ticks.append(g_completed_ga)
fig, ax = plt.subplots(
3,
1,
sharex=True,
height_ratios=[24, 1, 1],
figsize=(12, 48),
layout='constrained',
)
sns.heatmap(
gvm_df.sort_index(axis=1).T,
vmin=0.0,
vmax=1.0,
cmap='viridis',
cbar=True,
cbar_kws={'fraction': 0.01, 'anchor': (0.0, 1.0)},
ax=ax[0],
)
ax[0].set_title('Population average of gene values after each generation')
ax[0].axvline(x=best_generation_ga, color='C7')
ax[0].set_xlabel('generation')
ax[0].tick_params(axis='both', reset=True)
ax[0].set_xticks(x_ticks)
ax[0].set_xticklabels(x_ticks)
ax[1].set_xlabel('generation')
ax[1].plot(list(range(2, g_completed_ga + 1)), gene_maes[1:g_completed_ga])
ax[1].vlines(
x=best_generation_ga,
ymin=gene_maes[1:g_completed_ga].min(),
ymax=gene_maes[1:g_completed_ga].max(),
colors='C7',
)
ax[1].set_xlabel('generation')
ax[1].set_ylabel('MAE')
ax[1].set_title(
'Mean absolute error of average gene values between current generation and previous generation'
)
ax[1].tick_params(axis='both', reset=True)
ax[2].set_xlabel('generation')
ax[2].plot(
list(range(1, g_completed_ga + 1)),
best_objective_per_gen_ga[:g_completed_ga],
)
ax[2].vlines(
x=best_generation_ga,
ymin=min(best_objective_per_gen_ga[:g_completed_ga]),
ymax=max(best_objective_per_gen_ga[:g_completed_ga]),
colors='C7',
)
ax[2].set_xlabel('generation')
ax[2].set_ylabel('best objective')
ax[2].set_title('Best objective')
fig.suptitle('Genetic algorithm')
fig.savefig('ga-performance.png')
fig.show()best objective: 33705.569572544795
best generation: 787
objective runs: 600525
time to best: 158.027 sec
best features: ['BedroomAbvGr', 'BldgType_1Fam', 'BsmtFinSF1', 'BsmtFinSF2', 'BsmtUnfSF', 'Condition1_Norm', 'Condition2_PosN', 'ExterQual_Ex', 'Exterior1st_BrkFace', 'Functional_Sev', 'Functional_Typ', 'GarageArea', 'GrLivArea', 'HeatingQC_Ex', 'KitchenQual_Ex', 'LandContour_HLS', 'LotArea', 'LotConfig_CulDSac', 'LowQualFinSF', 'Neighborhood_BrkSide', 'Neighborhood_Crawfor', 'Neighborhood_NWAmes', 'Neighborhood_NoRidge', 'Neighborhood_NridgHt', 'Neighborhood_Somerst', 'Neighborhood_StoneBr', 'OverallCond', 'OverallQual', 'PoolArea', 'RoofMatl_WdShngl', 'SaleCondition_Normal', 'SaleType_New', 'TotalBsmtSF', 'WoodDeckSF', 'YearBuilt']上下滑动查看更多遗传算法-优化历史上面是一段优化算法的描述,该算法用于在1000代进化过程中寻找最佳特征组合。进化过程将特征的受欢迎程度用热图可视化表示,横向代表不同特征,纵向代表进化代数。热图上颜色越深,代表该特征在该代群体中越受欢迎。从热图可以看出,一些特征在整个进化过程中保持较高受欢迎度;另一些特征则很快被淘汰;还有些特征的受欢迎程度随着进化代数的增加而有所波动。该可视化展示了进化算法在特征选择过程中的动态变化,有助于了解算法的工作原理和特征的重要性。
各种方法之间的比较
我们尝试了三种不同的技术:SFS、CMA-ES 和 GA。就找到的最佳目标和找到目标所花费的时间而言,它们之间的比较如何?
这些测试是在一台安装了Ubuntu 22.04和Python 3.11.7的AMD Ryzen 7 5800X3D(8/16核)机器上进行的。SFS和GA分别通过一个包含16个Worker的多进程池来运行目标函数。CMA-ES是单进程的,多进程运行似乎没有明显的改进,但我相信如果有更多的工作来使算法并行化,情况会有所改善。
这些是执行时间。对SFS来说,是总执行时间。对于CMA-ES和GA,这是达到最佳解决方案所需的时间。时间越短越好。
SFS: 42.448 sec
GA: 158.027 sec
CMA-ES: 48.326 sec目标函数被调用的次数--越少越好:
SFS: 22791
GA: 600525
CMA-ES: 20000为目标函数找到的最佳值,与基准值相比——越少越好:
baseline BIC: 34570.1662
SFS: 33708.9860
GA: 33705.5696
CMA-ES: 33703.0705在目标函数方面,遗传算法(GA)能够取得比序列前向选择(SFS)更好的结果,因为它能够利用尽可能多的CPU内核来并行运行目标函数,但代价是它的运行速度是所有算法中最慢的。GA调用目标函数的次数远远超过其他方法。对超参数进行进一步优化可能会提高GA的性能。
相比之下,SFS的运行速度很快(能够在所有CPU内核上并行运行),但其性能一般。SFS是所有算法中最简单的。
如果你只是想快速估算出近似最优的特征集,而不太在意精度,SFS是一个不错的选择。
然而,如果你追求最优目标值,无约束进化策略(CMA-ES)似乎是最好的选择。