近年来,随着单细胞技术的迅速发展,我们得以测量了单个细胞的各种特征从而获取单细胞多模态数据(比如scRNA-seq,scATAC-seq,Patch-seq)。
这些数据有助于我们深入了解细胞功能和分子机制。比如研究人员近来多通过机器学习方法来分析单细胞多模态数据间的关系,进而理解细胞类型和疾病所涉及的生物学机制。
但是单细胞多模态数据的获取常常成本高昂,并且模态缺失时有发生。而现有的机器学习方法通常需要完全匹配的多模态数据才能进行数据填补和嵌入,不适用于模态缺失的情形。
为了解决这个问题,美国威斯康辛大学麦迪逊分校王岱峰实验室开发了一种基于联合变分自动编码器的开源机器学习方法——Joint Variational Autoencoders for Multimodal Imputation and Embedding(JAMIE)。
JAMIE可用于单细胞多模态数据整合分析,如数据对齐、嵌入,和对丢失数据进行添补,从而更好的预测细胞类型及功能。
此工作于近日发表于《自然–机器智能》(Nature Machine Intelligence)。

论文地址:https://www.nature.com/articles/s42256-023-00663-z
项目地址:https://github.com/daifengwanglab/JAMIE
JAMIE方法介绍
JAMIE训练了一种可重复使用的联合变分自编码器模型,将可用的多模态数据分别投影到相似的潜空间中,从而增强了单模态模式的推断能力。
如图1所示,为了执行跨模态填补,JAMIE将数据馈入编码器,然后将潜空间结果通过相反的解码器进行处理。
JAMIE将自编码器的可重复使用和灵活的潜空间生成与对齐方法的自动对应估计相结合,从而能够处理不完全对应的多模态数据。
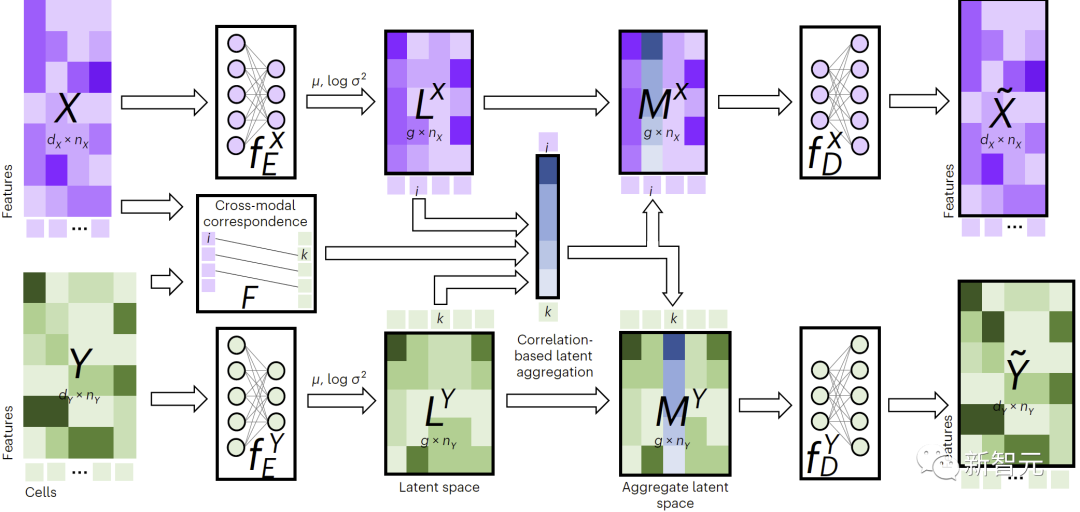
图1. JAMIE方法总览
具体而言,JAMIE可以分为以下两步:
- 输入数据预处理。以双模态为例,假设模态对应数据矩阵分别为和。注意这里特征维度和可以不同,样本数目和也可以不同。预处理对每个矩阵的每一行都归一化成均值0和方差1。如果有对应数据,用户可以提供模态相关矩阵来改进性能,其中 表示模态中的第个样本和模态中的第个样本完全对应,表示没有已知的对应关系,表示有部分的对应关系。
- 利用联合变分自编码器学习每个模态的相似潜空间: 和 ,其中(默认,用户可调节)是潜空间维度。训练过程中,JAMIE最小化如下损失函数:
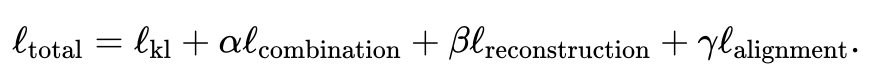
总损失函数包含四项。
其中第一项计算变分自编码器推断出的分布与多元标准正态分布之间的Kullback-Leibler (KL)散度,有助于保持潜空间的连续性;第二项强制对应样本的相似性;第三项是重构数据矩阵和原始数据矩阵之间的平均平方误差和;第四项利用推断的跨模态对应关系来调整生成的潜空间。
各项的具体表达方式见论文原文。第二、三、四项的相对第一项的权重可由用户自行调节,JAMIE也提供了可适用于常用情况的默认权重。
下述表格展示了JAMIE与当前最先进方法的模型和适用范围的对比。JAMIE将几种不同的整合和插补方法的特征统一到一个单一的架构中,因此能够进行缺失模态插值,从而具有非组学数据兼容性、且能处理只有部分对应关系的多模态数据的优点。
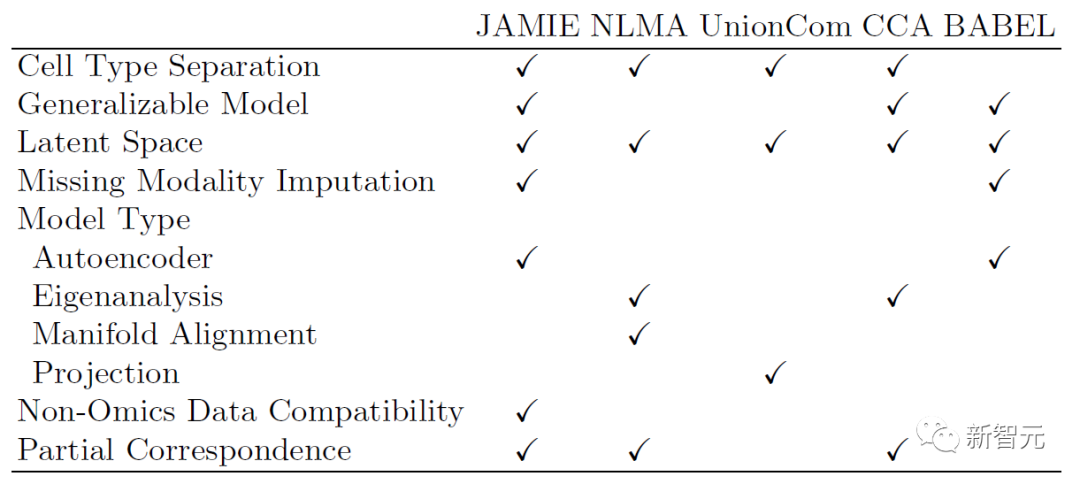
表1. 各种多模态整合和缺失模态填补方法的比较。JAMIE在一个单一的架构中统一了来自多种不同整合和插补方法的特征。NLMA:非线性流形对齐[15],UnionCom[7],CCA:典型相关分析[15, 16],BABEL[5]。
JAMIE的主要应用
多模态数据的整合和表型预测
对多模态数据的整合可以改进分类性能、增进对表型知识和复杂生物机制的理解。
给定两个数据集、和对应关系,JAMIE可以根据训练好的编码器和生成潜空间数据、,并基于、进行聚类或者分类。
基于潜空间数据的聚类具有几个优势,如将两种模态都纳入到特征生成。然后,JAMIE可以预测样本对应关系,并如细胞类型预测。
对于部分标注的数据集,同一聚类的细胞们应该具有相似的类型。
JAMIE在生成潜空间数据的过程中就进行了分离了不同类型数据的特征,因此通常不需要复杂的聚类或分类算法就可以达到较好的效果。
对于高维数据,JAMIE使用UMAP[32]进行细胞类型聚类可视化。
跨模态数据填补
目前跨模态填补的很多方法不能展示它们学习到了用于填补目的的潜在生物机制。
对比于前馈网络或线性回归方法,JAMIE能基于更严格的数学基础更好的学习到潜在的生物机制来预测缺失数据。
图2展示了JAMIE用于跨模态数据填补的流程。JAMIE先是针对训练数据训练编码和解码模型。
对于新数据 ,JAMIE首先利用数据学习到的编码器将其投影到潜空间得到 ,然后通过聚合潜空间特征的方法得到 ,最后通过对应的解码器将解码成缺失模式的数据。
JAMIE使用潜空间预测细胞的对应关系,这可能有助于理解数据特征和表型之间的关系。
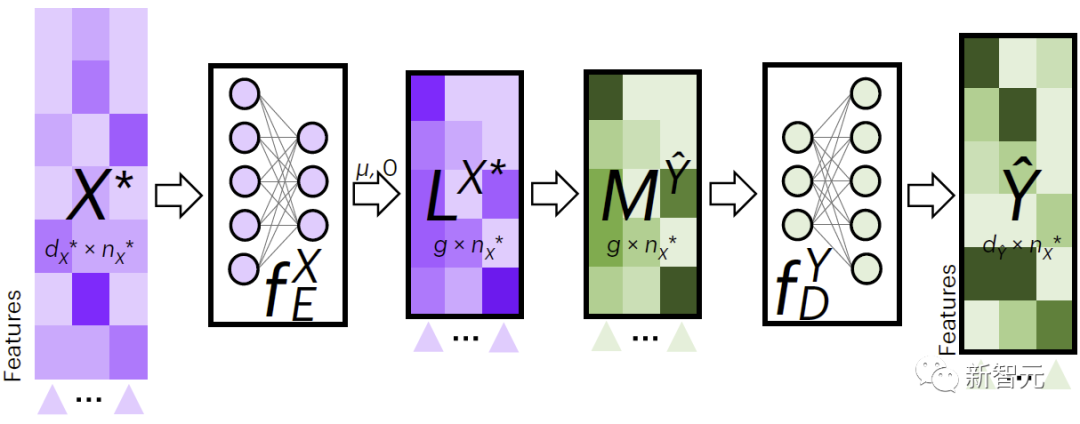
图2. JAMIE跨模态插补
潜空间特征和填补特征的解释
为了解释训练的模型,JAMIE采用了SHAP(SHapley Additive exPlanations)[18]。
SHAP通过对模型生成的个体预测进行样本调制来评估各个输入特征的重要性。这可以用于各种有趣的应用。
如果目标变量可以通过表型轻松分离,SHAP可以确定进一步研究的相关特征。此外,如果我们进行填补,SHAP可以揭示模型学到的跨模态联系。
给定模型和样本,学习到SHAP值,使得,其中是背景特征向量。
如果,则SHAP值的总和和背景输出将等于,其中每个与对模型输出的影响成比例。
另一种有用的技术是选择一个关键指标用于分类(例如,LTA[7,19])或填补(例如,填补特征与测量特征之间的对应关系),并在模型中逐个移除(用背景值替代)每个特征来评估该指标。
然后,如果关键指标变得更糟,这表明被移除的特征对于模型的结果更为重要。
实验结果
JAMIE采用了四个常用的单细胞多模态数据集进行验证。
(1)来自MMD-MA的分支流形的高斯分布采样生成的模拟多模态数据(300个样本,3个细胞类型);
(2)来自小鼠视觉皮层(3,654个样本,6个细胞类型)和小鼠运动皮层(1,208个样本,9个细胞类型)的单个神经元细胞的Patch-seq基因表达和电生理特征特征数据;
(3)来自人类发育中的大脑(21个孕周,覆盖人类大脑皮层的7种主要细胞类型)中8,981个样本的10x单细胞多组学基因表达和染色质可及性数据;
(4)来自COLO-320DM结肠腺癌细胞系的4,301个细胞的scRNA-seq基因表达和scATAC-seq染色质可及性数据。
评估发现,JAMIE明显优于其他方法(如图三的MMD-MA的分支流形模拟数据结果比较,和图四小鼠视觉皮层数据结果比较)并优先考虑了多模态填补的重要特征,同时在细胞分辨率层面上提供了潜在的新机制洞见。
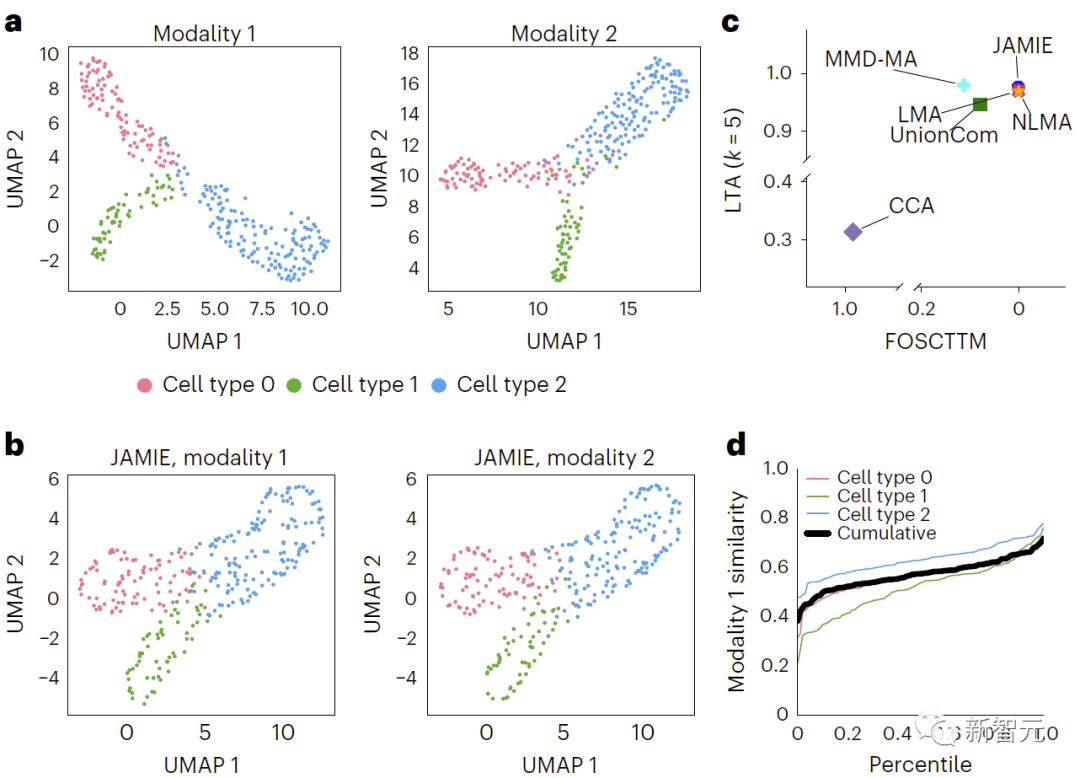
图3. 模拟多模态数据结果比较:a、原始空间的UMAP,按细胞类型上色。b、JAMIE潜在空间的UMAP。c、JAMIE和现有技术(CCA[15,16],LMA[15],MMD-MA[8],NLMA[15]和UnionCom[7])在使用所有可用的对应信息进行细胞类型分离时的比较。x轴为更接近真实均值的样本比例,y轴为LTA[7,19]值。d、模态1中测量值和插补值之间相似性(1-JS距离)的累积分布。黑线对应不同细胞类型的平均相似性,而每个彩线分别对应一个细胞类型的相似性。
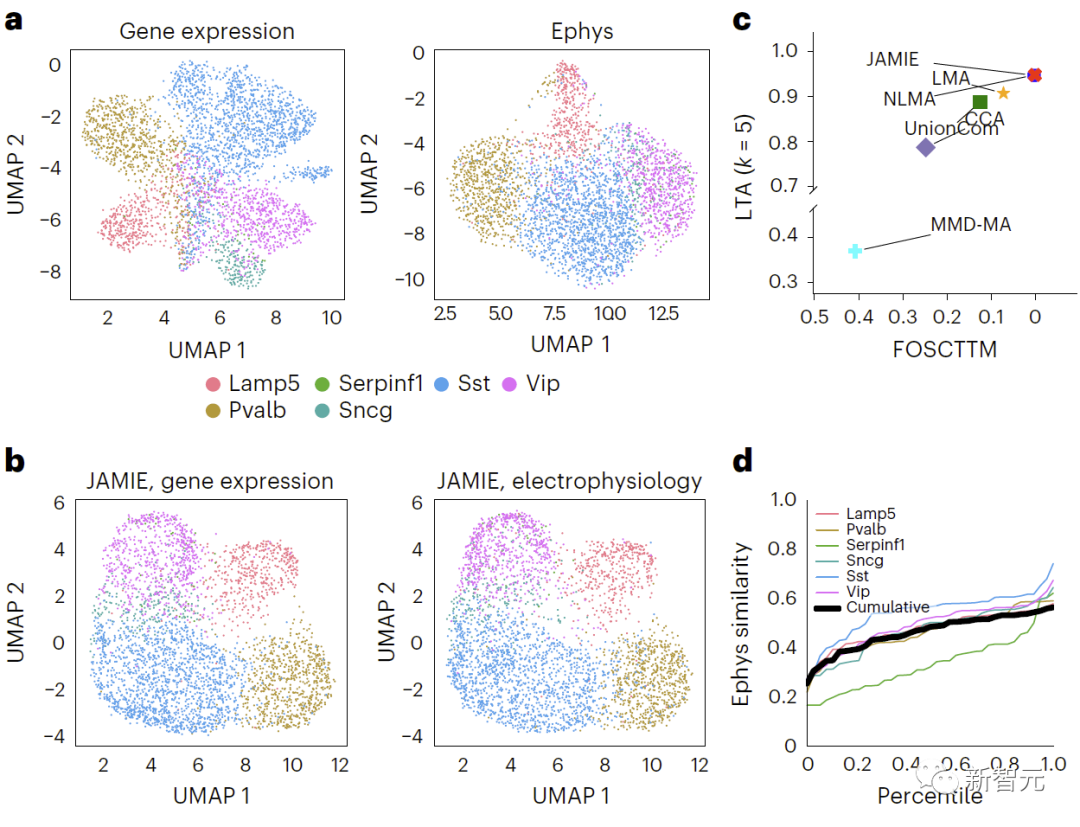
图4. 在小鼠视皮层中的基因表达和电生理特征结果比较:a、原始空间的UMAP,按细胞类型上色。b、JAMIE潜在空间的UMAP。c、JAMIE和现有技术(CCA[15,16],LMA[15],MMD-MA[8],NLMA[15]和UnionCom[7])在使用所有可用的对应信息进行细胞类型分离时的比较。x轴为更接近真实均值的样本比例,y轴为LTA[7,19]值。d、模态1中测量值和插补值之间相似性(1-JS距离)的累积分布。黑线对应不同细胞类型的平均相似性,而每个彩线分别对应一个细胞类型的相似性。
总结
总而言之,JAMIE 是一种用于单细胞多模态数据整合预测的新型深度神经网络模型。
它适用于复杂、混合或部分对应的多模态数据,通过一种依赖于联合变分自编码器(VAE)结构的新颖潜在嵌入聚合方法来实现。除了上述的优越性能外,JAMIE 还具有高效的计算能力和较低的内存使用需求。此外,预训练模型以及学习到的跨模态潜在嵌入可以在下游分析中进行重复使用。
当然对于较大的数据集,训练变分自编码器(VAEs)需要耗费大量时间。因此,JAMIE 中的自动 PCA 等先前特征选择方法有助于减轻时间要求。由于VAE使用重建损失,数据预处理也至关重要,以避免大量或重复的特征对低维嵌入特征产生不成比例的影响。对于特定的跨模态插补,必须仔细考虑训练数据集的多样性,以避免对最终模型产生偏差并对其泛化能力产生负面影响。JAMIE 还可以潜在地扩展到对来自不同来源而不是不同模态的数据集进行对齐,例如在不同条件下的基因表达数据。
作者介绍
论文作者Noah Cohen Kalafut(计算机系博士生),黄翔(高级研究员),王岱峰(PI)隶属于威斯康辛大学麦迪逊分校生物统计和医学信息学系、计算机科学系和威斯曼研究中心。通讯作者为王岱峰教授。
成立于1973年的威斯曼中心半世纪以来一直致力于推进人类发育,神经发育障碍和神经退行性疾病方面的研究。




































