分子表示学习在 AI 辅助药物发现研究中起着至关重要的作用。在传统药物研发中,常用的分子对接模型需要进行大量的构型采样与优化,并筛选出较为稳定的结构。这类策略效率较低,难以应用于高通量的蛋白质对接任务。本文介绍的基于分子表面黎曼流形的深度学习表示方法 (Harmonic Molecular Representation, HMR) 实现了更准确、高效的蛋白质对接模型开发。HMR 用二维黎曼流形建模分子表面,结合调合分析技术与神经网络实现流形上几何、化学信号的多尺度传播和两个蛋白质表面之间的匹配度比较,进而利用「蛋白质拼图」的逻辑实现蛋白质分子刚性对接 (rigid protein docking) 。实验表明,基于 HMR 的分子对接模型比当前深度学习 SOTA [1] 有更高的准确性,并且较传统分子对接方法提速 100 倍以上。相关论文已被收录于 ICLR 2023。
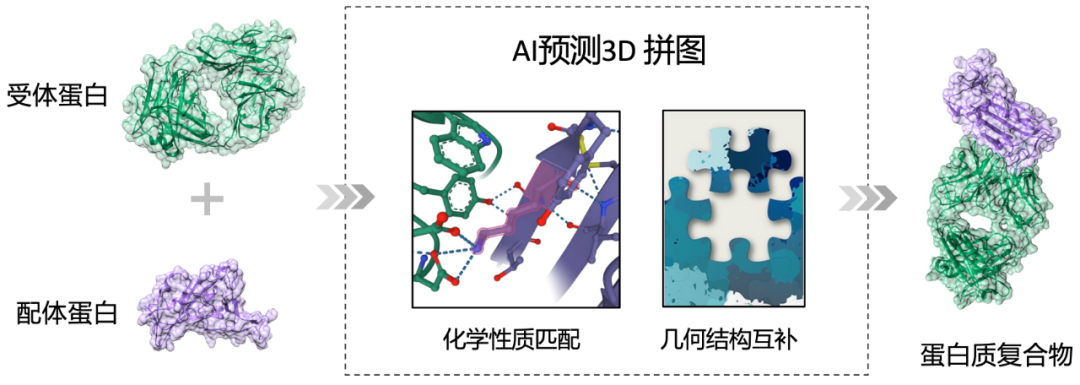
蛋白质之间的相互作用是实现其生物活性的重要基础,例如人体可以产生抗体蛋白(上图绿色部分)与入侵的病菌(紫色部分)结合从而抑制疾病。生物制药研究通过分析生物分子之间相互作用的物理、化学机制,进一步设计出可以与一些特定靶点相结合的新型药物分子(如研发新冠抗体)。在微观尺度下,蛋白质之间的相互结合主要由分子间作用力决定,例如氢键、静电力、疏水作用等。
在传统药物研发中,分子对接技术 (molecular docking) 通过物理方法建模这些微观作用来模拟计算真实生物体内两个分子相互作用的稳定结构。这些传统的分子对接模型需要进行大量的构型采样及优化,并筛选出其中较为稳定的结构作为预测结果。这种基于采样和筛选的策略导致传统方法效率较低,难以应用于高通量的蛋白质对接任务(例如从 1 万个蛋白中筛选出最有可能与病菌表面结合的 10 个来进行湿实验测试)。研发准确且高效的分子对接模型可以帮助快速筛选出适合进行湿实验测试的蛋白质分子,从而提高新药研发的效率。
为了开发更加准确且高效的蛋白质对接模型,字节跳动 ByteDance Research 团队设计了一套基于分子表面的几何深度学习方案。该方案的核心思想是训练 AI 模型从拼图的角度去理解蛋白质之间的相互作用,并按照拼图的思路实现蛋白质复合物构型预测。

- 论文地址:https://openreview.net/pdf?id=ySCL-NG_I3
- 代码地址:https://github.com/GeomMolDesign/HMR
具体来说,如果两个蛋白质可以结合,那么其结合区域必须同时满足化学性质匹配和几何结构互补这两个条件(下图)。因此直观上可以将其类比为一个拼图问题:只要能够在蛋白质表面分别找到两块形状和纹理都能匹配的「拼图」,那么就可以将这两个蛋白质拼在一起形成稳定的蛋白质复合物。
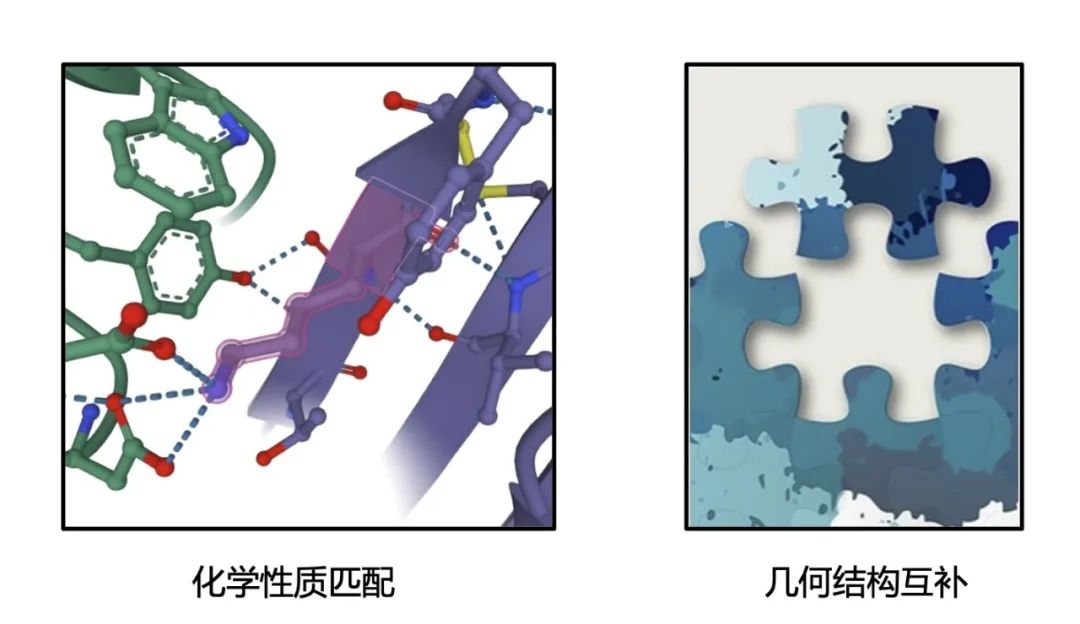
图:能够结合的蛋白质分子必须同时满足化学性质匹配和几何结构互补这两个条件,类似于拼图问题。
基于上述假设,团队提出了一种基于分子表面黎曼流形的深度学习表示方法 (Harmonic Molecular Representation, HMR) :用二维黎曼流形建模分子表面,结合调合分析技术与神经网络实现流形上几何、化学信号的多尺度传播和两个蛋白质表面之间的匹配度比较,进而利用「蛋白质拼图」的逻辑实现蛋白质分子刚性对接 (rigid protein docking) 。直观上,神经网络模型需要从大量蛋白质复合物的结构(训练集)中学习这类拼图的几何、化学规律,从而可以去预测没有见过的(测试集)蛋白质复合物结构。
实验表明,基于 HMR 的分子对接模型比当前深度学习 SOTA [1] 有更高的准确性,并且较传统分子对接方法提速 100 倍以上。接下来的 3 个章节将分别介绍黎曼流形分子表示、分子表面几何深度学习和蛋白质拼图模型的主要思想和技术概要。
§1 基于黎曼流形的分子表示
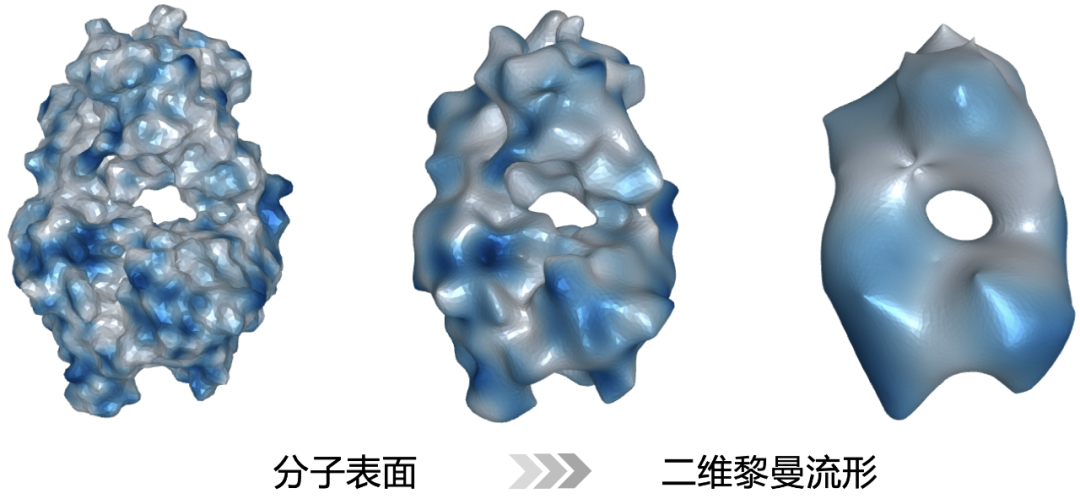
图:抗体蛋白 (PDB ID: 6W41) 表面流形在不同分辨率下的表示。颜色代表分子表面静电势能函数,蓝色为负电荷区域。
生物分子的表面通常是指该分子在溶液中与溶剂(例如水分子)形成的界面。我们可以将这样的界面表示为一个三维空间中的二维黎曼流形 (Riemannian manifold)。上图展示了一个抗体蛋白在不同分辨率下的表面流形结构,流形上的颜色对应分子表面的静电势能函数分布。换句话说,流形的结构勾勒了分子的形状,而流形上分布的函数则可以表示分子表面的化学性质。因此,利用黎曼流形可以统一地表示分子的几何结构和化学性质,从而整合分子信息并帮助 AI 模型更好地学习蛋白质的结构 - 活性关系。
在黎曼流形的表示下,每个分子都有一套独特的「形状基因」(Shape-DNA) 。这些形状基因被定义为分子表面流形的拉普拉斯-贝尔特拉米算子 (Laplace-Beltrami Operator, LBO) 的本征值的集合 {𝝀ᵢ},它们提供了一种描述分子形状的内蕴方法,不受三维空间中分子的位置和取向影响。
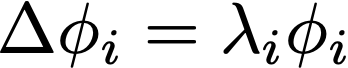
图:黎曼流形上的拉普拉斯特征方程。Δ 代表拉普拉斯-贝尔特拉米算子,φᵢ和𝝀ᵢ分别代表第 i 个本征函数和对应的本征值。不同的分子表面流形有其独特的特征方程的解。
这些形状基因所对应的 LBO 本征函数 {φᵢ} 构成了分子表面流形上的一组(标准正交的)基函数 (orthonormal basis functions)。我们可以简单将它们类比为正弦 / 余弦函数组成的傅立叶基组在黎曼流形上的拓展。因此,这些形状基因和基函数可以帮助我们在分子表面进行调和分析 (harmonic analysis),也就是将一个流形上的函数展开成一系列基函数的线性组合的形式(下图)。对于同一个分子表面,不同的化学性质(如疏水性、静电势能)可以非常简洁地表示为一组线性组合系数(一维数组 [c₀, c₁, c₂,...])。
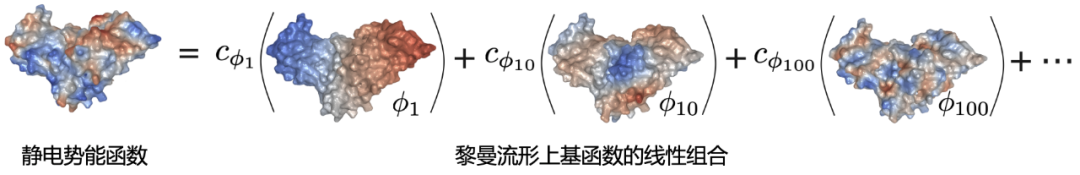
图:分子表面黎曼流形上的调和分析。可以将流形上的函数展开成一系列基函数线性组合的形式。仅选择性展示了 3 个基函数,其实所有的展开项都参与线性组合。
这种黎曼流形的表示是对分子表面的直接建模,得到的形状基因和 LBO 基函数受不同离散化建模方式的影响较低(例如分子表面预处理过程中的离散采样密度和三角剖分方式 [2]),因此增加了建模的鲁棒性。
§2 分子表面几何深度学习
上一章主要介绍了文章如何利用黎曼流形建模分子表面的几何结构(对应拼图的形状),这一章我们简要描述如何训练神经网络学习分子表面的化学性质(对应拼图上的纹理)。
利用分子表面的形状基因和 LBO 基函数来建模分子的几何、化学性质的方法为我们提供了一种新的分子表示学习思路。基于表面的分子建模专注于对分子外部特性的描述,在一些下游任务上(比如蛋白质相互作用)可能比基于氨基酸或全原子建模的三维图神经网络 (Euclidean graph neural networks) 更具有优势。「形状基因」是对分子表面整体进行不同频率(或粒度)信号的建模,不需要提前对表面区域进行预切割处理 [2],也让模型适合学习不同尺度 (multi-scale) 的分子信息。
基于这套思路,团队设计了一套几何深度学习的方案用于分子表面上的全局信息传递,从而帮助神经网络学习到更丰富的分子表面几何、化学性质表示。文中提出了流形调和信息传递 (manifold harmonic message passing) 的新方法:一种类似于热扩散的流形表面信息传递机制,但建模上更为灵活。热扩散机制可以看成是对信号进行低通滤波操作,因此低频信号比高频信号更容易被传播开 [3];而流形调和信息传递允许对不同频率的波段进行过滤,实现高频信号的独立传播 [4],因此传播距离更远,表示粒度更细。结合残差连接等神经网络结构,文中提出的方法可以学习分子表面不同尺度、不同距离的几何与化学信息,从而对分子有更好的建模能力。
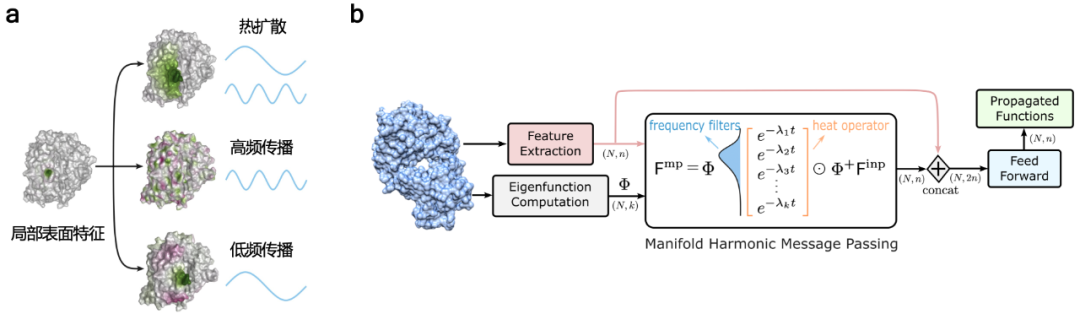
图:利用流形调和信息传递机制进行分子表面的表示学习。(a)分子表面的局部特征可以通过不同的信息传递机制在流形上传递,实现有效的几何、化学性质表示。(b)流形上的信息传递神经网络结构。
§3 蛋白质拼图 AI 模型
现在,我们有了(§1)基于黎曼流形的分子几何表示方法和(§2)神经网络学习到的分子表面化学性质函数这两种工具,接下来可以利用它们来解决蛋白质复合物的构型预测问题。
具体而言,给定受体蛋白和配体蛋白的结构,我们希望预测出它们结合后复合物的构型。这里主要涉及两个子问题:(1)结合位点在哪里;(2)受体 - 配体以什么样的空间姿态相结合。文中提出,蛋白质之间的结合位点应该具备几何结构互补和化学匹配这两个重要条件,因而蛋白质复合物构型预测可以看成是一种「蛋白质拼图」问题。类似于人类解决拼图的思路:首先找到两块拼图之间的拼接面(预测结合位点),再利用形状和花纹的相似性旋转其中一块拼图放置到正确的位置(分子对接)。
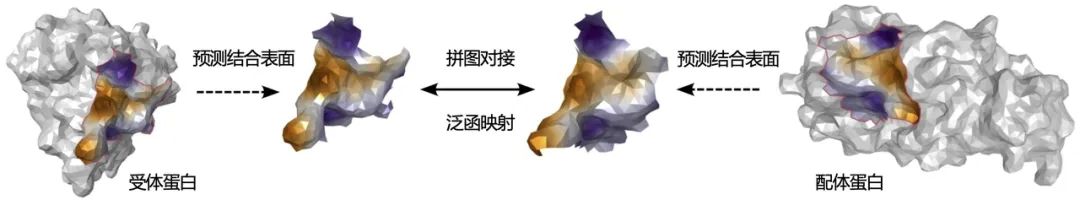
图:「蛋白质拼图」流程示意图:模型首先预测两个蛋白质之间的结合表面区域,再利用神经网络所学函数的匹配性实现高选择性的拼图。
在具体的模型构建上,团队首先利用文中提出的 HMR 模块进行表面位点的预测 (binding site prediction)。通过流形信息传递机制实现分子表面的特征学习,并引入交叉注意力机制 (cross attention) 实现蛋白质分子间的信息交流。该模块最终的输出是模型对分子表面的某个区域是否属于蛋白结合位点的二元预测。可以理解为这一步对应的是寻找拼图上缺失的一块。
接下来进入分子对接模块:预测得到的蛋白结合表面上应具有一定的函数对应关系 (functional correspondence),因为分子间相互作用是由匹配的化学作用(如静电力和氢键)决定的。也就是说能够匹配的拼图上所对应的纹理也应该有一致性。根据这样的假设,团队进一步利用泛函映射技术 (functional map) 将这种函数对应关系转化为受体 - 配体结合位点之间的空间对应关系,并使用 Kabsch 算法将配体分子进行空间上的旋转和平移,最终得到对接后的蛋白质复合物结构。
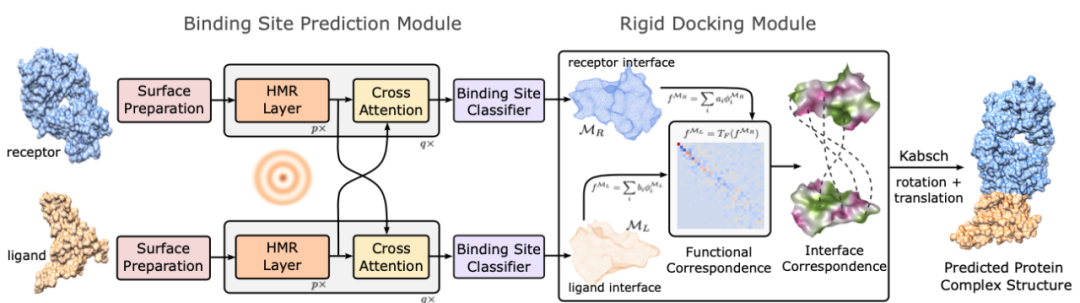
图:「蛋白质拼图」模型流程图,主要分为结合表面预测(左👈)和刚性分子对接(右👉)两个模块。
文中将这种「蛋白质拼图」方法和三维图神经网络的方法 EquiDock [1] 以及基于「采样 - 筛选」策略的传统方法(ATTRACT 和 HDOCK)进行了比较。在蛋白质对接标准测试集 Docking Benchmark 5.5 上,「蛋白质拼图」的方法在各个衡量项目上均比三维图神经网络取得了更好的效果,甚至接近于一些传统方法。而这种基于深度学习的方法预测分子对接的速度比传统方法提高了 100 倍以上。
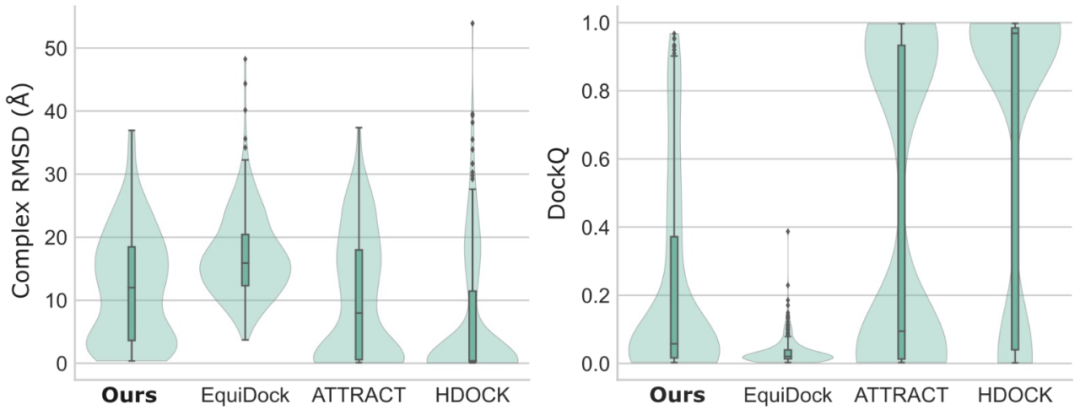
图:「蛋白质拼图」模型(显示为 ours)和基于三维图神经网络的深度学习模型 SOTA (EquiDock) 以及传统刚性分子对接模型 ATTRACT 和 HDOCK 的比较。Complex RMSD 越小、DockQ 越高说明预测的蛋白质复合物结构越接近真实结构。
§4 总结
这项工作提出了一种新的基于分子表面的深度学习建模方式,利用黎曼流形和形状基因对化学、物理以及几何相关的性质进行建模,并在蛋白质分子刚性对接的任务上超越基于三维图神经网络的深度学习方法。总结来说:
- - 不同于常见的基于序列或者二维 / 三维图结构的建模方式,团队采用了基于表面的分子建模方法。这种建模思路既保留了分子整体的三维结构,又避免了对于大分子内部结构的冗余建模。在和蛋白质功能、蛋白质相互作用等表面相关的下游任务中可能更具优势。
- - 采用黎曼流形直接对分子表面进行建模,并用分子形状基因和对应的本征函数来表示表面的函数分布。这种建模方法不受表面离散采样和三角剖分方法的影响,因而具有更好的鲁棒性。团队同时展示了基于调和分析和滤波概念的表面传播深度学习模块,以及基于泛函映射的表面对接方法。
- - 文中利用这种表面建模方法和「拼图」的思想实现了基于深度学习模型的蛋白质刚性分子对接。相比于目前基于三维图神经网络的深度学习模型取得了更好的效果,在速度上相比于传统方法也有大幅提升。