为什么会有 BioArchLinux 项目?
以目前科学相关的发行版为例,它们大多基于 Ubuntu ,比如 Bio-Linux 以及 Poseidon Linux ;也有基于 CentOS 或者 RHEL 的,比如 Scientific Linux 。但是最终这些发行版都慢慢不再活跃,Scientific Linux 发出的各种 公告 也是表现出身不由己。
从 Scientific Linux 的经历可以看出,如果将各种软件包打包在一个依赖商业公司或者由商业公司主导的发行版上,发展方向就会变得不可知,最初的目标和规划自然不能得以实现。最初 Scientific Linux 依赖付费的 Linux 发行版 RHEL ,后期依赖商业公司的免费社群发行版 CentOS(CentOS 8 以及之前是一个稳定的发行版),红帽将 CentOS 8 的生命周期草草结束,进而支持滚动发行版 CentOS Stream(现在是 RHEL 的上游发行版),因此 Scientific Linux 不得不变成基于 CentOS Stream 的发行版。只能说,Scientific Linux 一开始就选择错了。
再从 Bio-Linux 的角度来看待,Bio-Linux 本质上是把各类软件包打包到 Ubuntu 内之后形成的一个发行版。这必然有一个周期,在这个一年或者两年的周期内,各类软件总会有更新的,而 Bio-Linux 不考虑这个问题,所以会出现使用过时的版本来分析数据的情况,很明显这不利于研究。而且, Bio-Linux 8 自 2014 年发布了基于 Ubuntu 14.04 LTS 的发行版之后,就没在发行新的版本了,而目前 Ubuntu 22.04 LTS 都已经出来了。Bio-Linux 的 软件包 除了老旧,还特别冗杂,我需要的包他们不全有,我不需要的包他们有很多,这毫无疑问增加了我 PC 的负担。
Poseidon Linux 也有着类似的问题。这种发布发行版的方式滞后且需要重装系统,特别不方便。实在不如直接经营一个各类包的仓库,可以快速更新,不必频繁发布 ISO 文件又可以将软件更新到最新版。
所以,如果你希望想长期使用,那么就建议使用非商业公司关联的 Linux 系统;如果你需要参与 Linux 的发展,那么你就要寻找一个方便使用第三方仓库/官方仓库、且非商业公司关联的发行版。这里我们就选择了 Arch Linux。
同时,我们也不希望只是一群为 AUR 做贡献人,因为曾经我自己的设想是这个团体可以像 RedHat 那些发行版之类的 SIG,但是 SIG
的运作模式是为官方仓库贡献包。而 AUR 只是存储一个脚本,并不是一个预先编译好的包。这样带来的麻烦有很多,首先是 AUR
不能和官方仓库的包有冲突,但是这对于生物信息的目标用户群体是个麻烦事情,比如我要找 picard,但是 community 仓库里的 picard 已经是别的同名软件了,但是它只是在 community 仓库里,我不会用到它,因此我要几经周折地找到 AUR 里的 picard-tools。AUR
另外一个不方便的点在于软件包的来源不一定不被封锁,曾经我向我师姐十分热情的推销 Arch 系的发行版,她也觉得蛮好用,但当她想从 AUR
里下载软件时候,互联网限制了她的想象。但是,当我们组成了一个有镜像源的仓库的时候,我们就不需要担心这个问题了,来自互联网封锁国家的人们就无需忍受缓慢的互联网速度和法律风险访问他们所需要的软件了。
如何使用 BioArchLinux?
首先,BioArchLinux 本身的属性决定了用户可以在哪些地方使用它。 BioArchLinux 是一个生物工作者的 Arch Linux 社区,包含了一个生物学软件的 Arch Linux 存储库、可以编辑的 wiki 以及 Matrix 聊天频道。
在 Arch Linux 中使用 BioArchLinux
正如它本身的属性所定义,它可以用于 Arch Linux 及其衍生发行版(不包括 Manjaro stable & testing),从 BioArchLinux 安装软件很容易。只需几个简单的命令即可安装所需的软件包。
在最初接触 Linux 时候,我使用 Ubuntu 。当我想要安装生信软件的时候,我曾经一下午都在处理循环依赖的问题。这或许是某些发行版的特性,而且由于我是图形化安装的,我其实对未来怎么迁移系统并没有足够的把握。对于小白来说,好不容易装好的环境想要迁移很难避免重复性的工作。Arch Linux 的特性避免了这里很多问题,从打包的粒度考虑,循环依赖可以说是很罕见的了。另外就是当你需要构建一个包,你只需要会写 Shell 脚本再看一看维基,事情会容易很多。相比于 Debian 等发行版,这样其实会有利于你迁移你安装的软件。
当然,和其他软件仓库不同的是,BioArchLinux 仓库在可能的情况下,在每个包描述中提供了一个 DOI。 这使用户能够轻松地了解有关每个包的用途和方法的更多信息,并在准备出版物时快速识别适当的引用。
在 WSL 中使用 BioArchLinux
另外,当 Windows 和 macOS 用户需要使用 Linux 环境来运行生物信息软件的时候,也可以轻松使用 BioArchLinux。因为 BioArchLinux 同样提供 WSL 以及 Docker 镜像。
对于 Windows 用户优先推荐 WSL,因为 Docker 在 Windows 下依赖 WSL。只需要在任意一个镜像站点的 wsl 文件夹下找到 tar 文件即可。解压它,在安装了 wsl 的前提下双击 BioArch.exe 文件,就可以开始成功安装,安装好后键入下述命令即可进入:
在使用前需要做一些初始化的任务,比如初始化 WSL,这里的镜像地址可以更改为你喜欢的镜像,镜像列表见 mirrorlist 仓库 里的 mirrorlist.bio。
此时,你就可以使用该 WSL 了。
在 Docker 中使用 BioArchLinux
至于 Docker 的使用和 WSL 类似,只不过在安装完 Docker 后使用如下命令进入。进入后依然需要使用 WSL 初始化的命令初始 Docker 容器。
BioArchLinux 如何运作?
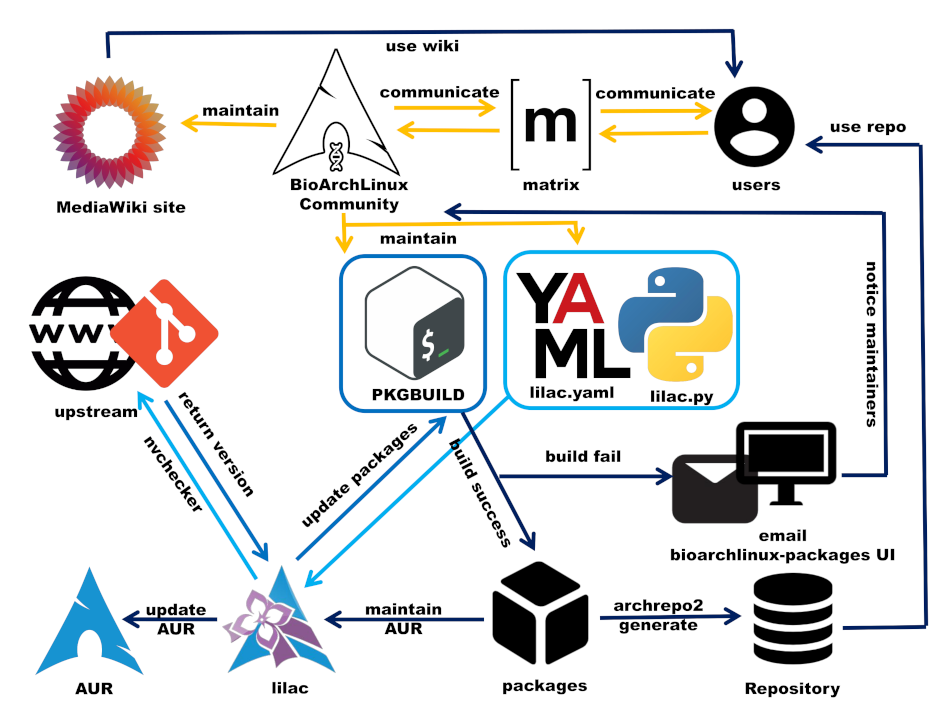
BioArchLinux 运行流程
BioArchLinux 存储库由几个开源软件包维护。 主要工具是一个名为 lilac 的 python 应用程序。
最基本的步骤是按照 Arch Linux 和 lilac.yaml 的标准编写脚本。我们编写一个 PKGBUILD shell 脚本和一个 YAML 文件(以及可选的 Python 脚本),并将它们放在 Git 存储库的一个文件夹中。
nvchecker 读取 lilac.yaml,获取上游网站的信息,可以查看最新版本。如果 nvchecker 无法从上游网站找到包版本,它会向管理员发送电子邮件报告问题。
nvchecker 的信息发送给 lilac,由 lilac 判断包是否需要升级。如果软件包需要升级,lilac 会将软件包发送到 Arch Linux 打包工具 devtools。
devtools 为软件包提供了一个干净的环境,只有 PKGBUILD shell 脚本中的依赖项列表允许构建。这可以避免在使用过程中丢失依赖项。如果包构建失败,则会自动向包维护者发送警告电子邮件。如果包构建成功,archrepo2 会将 Arch Linux 包放入特定路径,并生成一个新的数据库文件,形成一个全新的包仓库。如果 lilac.yaml 中含有维护 AUR 的指令,包更新也将退送给 AUR。
整个构建过程被记录为日志文件,可以使用 Rust 应用程序 bioarchlinux-packages 读取,并显示在日志网站上。
我们的维基网站是基于 MediaWiki 构建的。所有人都可以自由地为本网站贡献关于生物信息学软件的使用以及生物信息学概念和术语。
BioArchLinux 展望
上面讲了那么多的好,其实 BioArchLinux 也有很多的不足。
先从仓库说起,我们虽然在短短一年内有了约 4.2 k 的软件包,维护了约 4.7% AUR 包,但是,我们相比于 Debian Med 以及 bioconda 都有很大的数量上的差距,急需更多的维护者参与进来,并且需要不断提升打包的质量。
除此之外,比较急切的是我们国内镜像源目前仅仅有几家高校,南京大学、西安交通大学、山东大学以及南京邮电大学,我们希望更多的镜像站能够添加我们。另外因为计算机资源的问题,我们也没有 archive 网站,这给回滚造成了一定程度的困难。
其余就是扩大仓库的受众和加强社区的维护。虽然我们有了 WSL 还有 Dokcer,但是有些人很喜欢在虚拟机里运行,我们却提供不了 ISO 文件,也需要相关的维护人员。我们也没有专门的维基管理人员,有段时间因为没有限制用户注册,网站有被垃圾信息灌爆。
甚至我们在网站的搭建上面还是有欠缺,比如没有像 Arch Linux 那样的搜包界面,这需要更多开发人员的参与。除此之外,如何以非 root 用户的角色使用仓库仍然是一个很大的课题。
我们十分欢迎更多的人参与到我们的社区中来,一起做一些疯狂且美好的事情,不管再多困难,我相信,这个那么 FFF(community friendly, user friendly, earth friendly)的项目会长命百岁。(注:community friendly 帮助维护 Arch Linux community 的 AUR 软件包;user friendly 易于使用、以用户为中心;earth friendly 减少大家编译的次数,尽可能减少计算机资源的消耗。)
致谢
非常感谢 xTom、Mick Elliot 以及 Bipin Kumar 对这个项目的资助,也十分感谢一起为仓库工作的所有 BioArchLinux 成员。另外特别感谢 Arch Linux CN 依云 以及 imlonghao,没了他们维护的软件,BioArchLinux 不可能那么顺利的运作。同时也感谢南京大学、西安交通大学、山东大学以及南京邮电大学和其他为 BioArchLinux 提供镜像的机构和个人。最后,感谢之前 Bioinformatics Open Source Conference(BOSC)为参会免除会议费用。