不同于蛋白质领域,RNA 领域的研究往往缺少充足的标注数据,比如 3D 数据只有 1000 多个 RNA。这极大限制了机器学习方法在 RNA 结构功能预测任务中的开发。
为了弥补标注数据的不足,本文展示了一项可为 RNA 各类研究提供丰富结构功能知识的基石模型 ——RNA foundation model (RNA-FM)。作为全球首个基于 23 million 的无标签 RNA 序列通过无监督方式训练得到的 RNA 基石模型,RNA-FM 挖掘出了 RNA 序列中蕴含的进化和结构模式。
值得注意的是,RNA-FM 仅需要配比简单的下游模型,或是仅提供 embedding,就能够在很多下游任务中获得远超 SOTA 的表现,比如在二级结构预测中可以提升 20%,距离图预测可以提升 30%。大规模的实验证明,该模型具有极强的泛化性,甚至可以用于 COVID-19 以及 mRNA 的调控片段。

- 论文预印本:https://arxiv.org/abs/2204.00300
- 代码和模型:https://github.com/ml4bio/RNA-FM
- Server: https://proj.cse.cuhk.edu.hk/rnafm
引言
近年来,基于深度学习的生物计算方法在蛋白质领域取得了突破性的进展,其中最著名的里程碑当属谷歌 DeepMind 团队研发的端到端蛋白质 3D 结构预测框架 AlphaFold2。然而蛋白质只是诸多生物分子的一种,基因(DNA/RNA)作为蛋白质的产生源头,其相比于后者蕴含了更多的基础信息,有着更重要的研究价值。
一般而言,蛋白质是由用于编码(coding)的 RNA,也就是 mRNA,翻译得到的产物,一段固定的 mRNA 可以翻译为一段固定的蛋白质序列。而实际上这部分用于编码的 RNA 只占所有 RNA 序列的 2%,剩下的 98% 是非编码 RNA(non-coding RNA,ncRNA)。虽然 ncRNA 并不直接 “翻译” 成蛋白质,但是他们会折叠成具有特定功能的三级结构,在 mRNA 的翻译过程中或是其他生物机能中起到调控的作用。因此,分析 ncRNA 的结构以及功能是比蛋白质分析更为基础,也更为复杂的研究。
不过相比于计算方法较为成熟的蛋白质领域,目前基于 RNA 的结构和功能预测还处于初期,而原本适用于蛋白领域的计算方法也很难直接迁移到 RNA 领域。限制这些计算方法的主要是 RNA 数据的标注通常获取很难,需要耗费很多的实验资源和时间才能完成少量数据的标注,而计算方法大多又需要大量的标注数据进行监督才能发挥高性能。虽然有标注的数据不多,但 RNA 领域其实也积累了很多的无标注序列数据。本文的方法便是利用这些无标签的数据为各种下游任务提供额外的有效信息。
基于这种考虑,港中文、MIT、复旦及上海人工智能实验室团队提出了一个以无监督方式在 23million 的无标签纯 RNA 序列上训练的基石模型RNA foundation model (RNA-FM)。虽然数据在训练过程中没有提供标注信息,但是 RNA-FM 仍以无监督的方式挖掘出了这些 RNA 序列蕴含着的进化和结构模式。
如果能够有效地将 RNA-FM 应用于下游的 RNA 结构和功能预测任务中,这些计算方法必将受益于 RNA-FM 归纳得到的知识,进而实现性能表现上的提升。RNA-FM 的上游预训练以及下游的迁移和应用框架如下图所示。
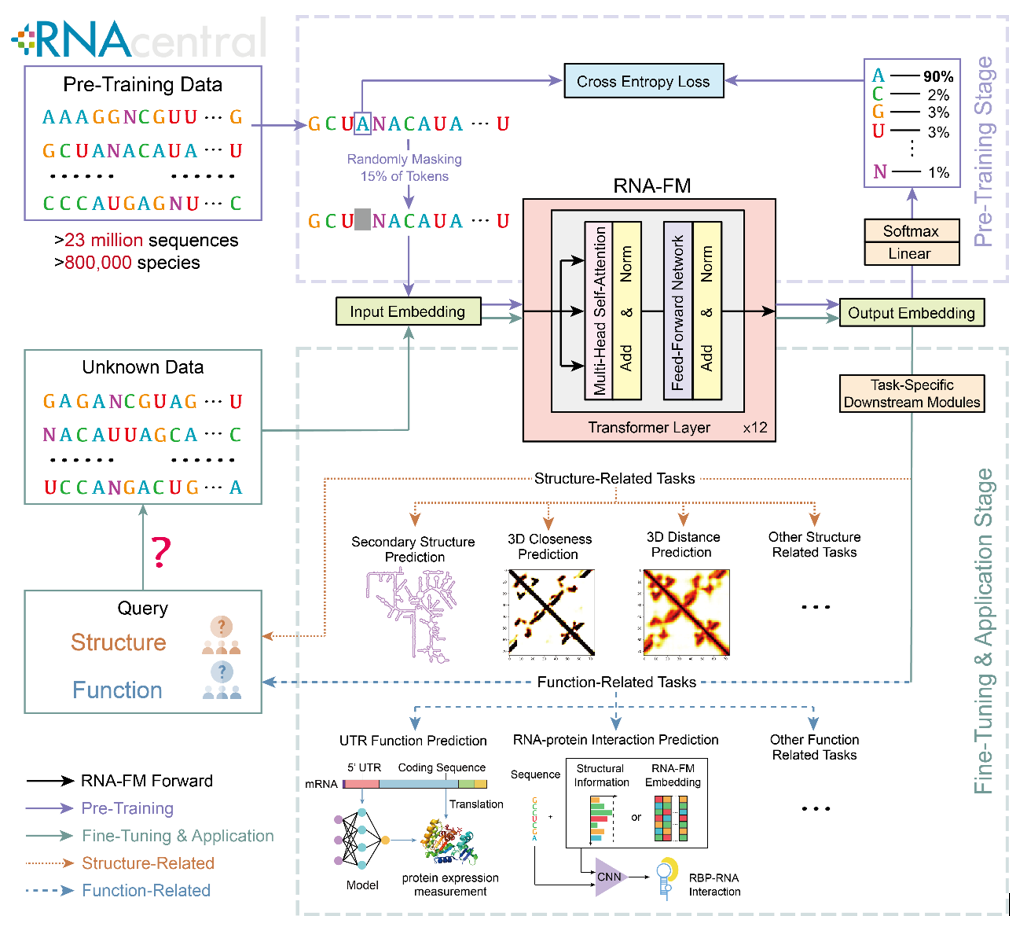
研究概览
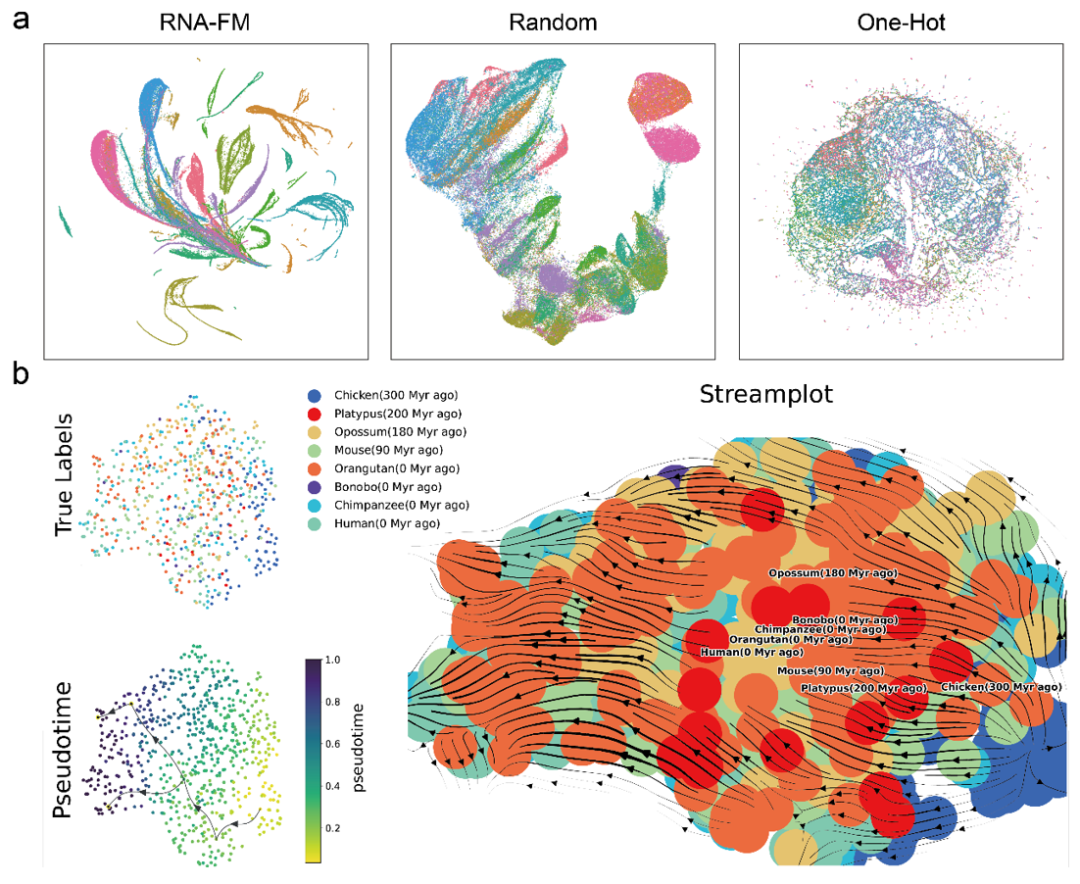
为了确认预训练的 RNA-FM 是否从大量的无标签数据中学到了 “知识” 以及学到了怎样的 “知识”,文章对 embedding 进行了一系列的分析。
首先是直接通过 UMAP 对各种特征进行简单聚类比较,发现来自预训练 RNA-FM 的 embedding 比其他 embedding 形成了具有更加明显的 RNA 种类聚落。这意味着 RNA-FM 的 embedding 确实包含了用于区分 RNA 种类的结构或功能信息。
接着,文章还利用轨迹推断(Trajectory inference)通过 RNA-FM embedding 去预测来自不同物种的 lncRNA 的演化。从下图 streamplot 上看,预测的物种之间演化的伪时间大致与真实的物种演化信息一致,说明 RNA-FM embedding 还包含一部分进化信息。
值得注意的是,无论是 RNA 种类的群落信息还是 lncRNA 的演化信息,RNA-FM 在训练中都没有直接接触过这些的标签。RNA-FM 完全是以自监督的方式仅从纯序列中发掘出了与结构、功能以及演化相关的模式。
更多实验结果
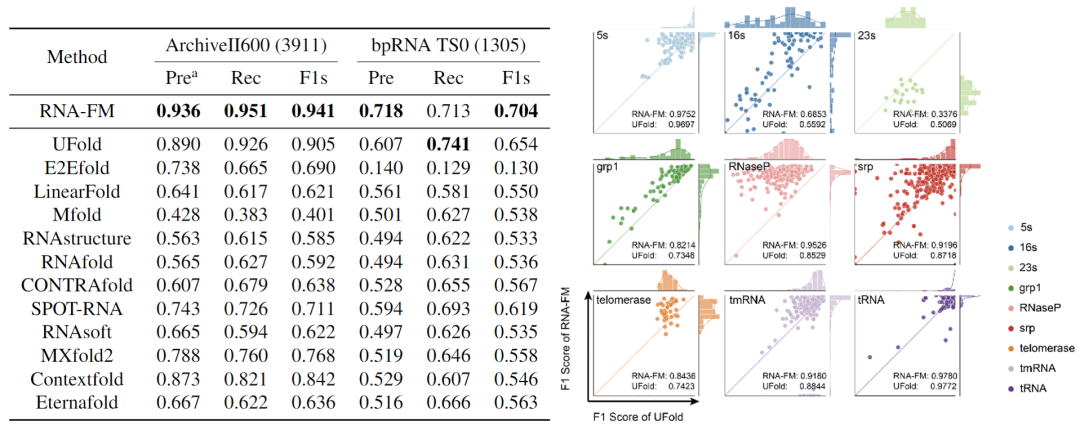
除了直接对 RNA-FM 的 embedding 进行分析,文章还尝试将 RNA-FM 引入到各种各样的下游 RNA 结构预测任务,包括二级结构、接触预测,距离预测,以及三级结构预测,且都取得了明显的提升。
尤其是在二级结构预测上,文章以 RNA-FM 作为主干,仅以一个简单的 ResNet 网络作为下游模型,就在两个公开数据集上超过了其他 12 种 state-of-the-art 方法,在 F1score 上优于其中最好的 UFold 达 3-5 百分点,在与 UFold 的 head-to-head 比较中,RNA-FM 在绝大部分的 RNA 类别上都超过 UFold。如果将 RNA-FM 和 E2Efold 结合,还可以进一步带来 5% 的表现提升。
为了验证模型的实际应用价值,文章利用 RNA-FM 对 COVID-19 数据进行完善的分析,包括利用 RNA-FM 精准预测 COVID-19 参照基因组(29870 nt)中关键调控元件,以及利用 RNA-FM embedding 粗略预测 COVID-19 主要变种的演化趋势。 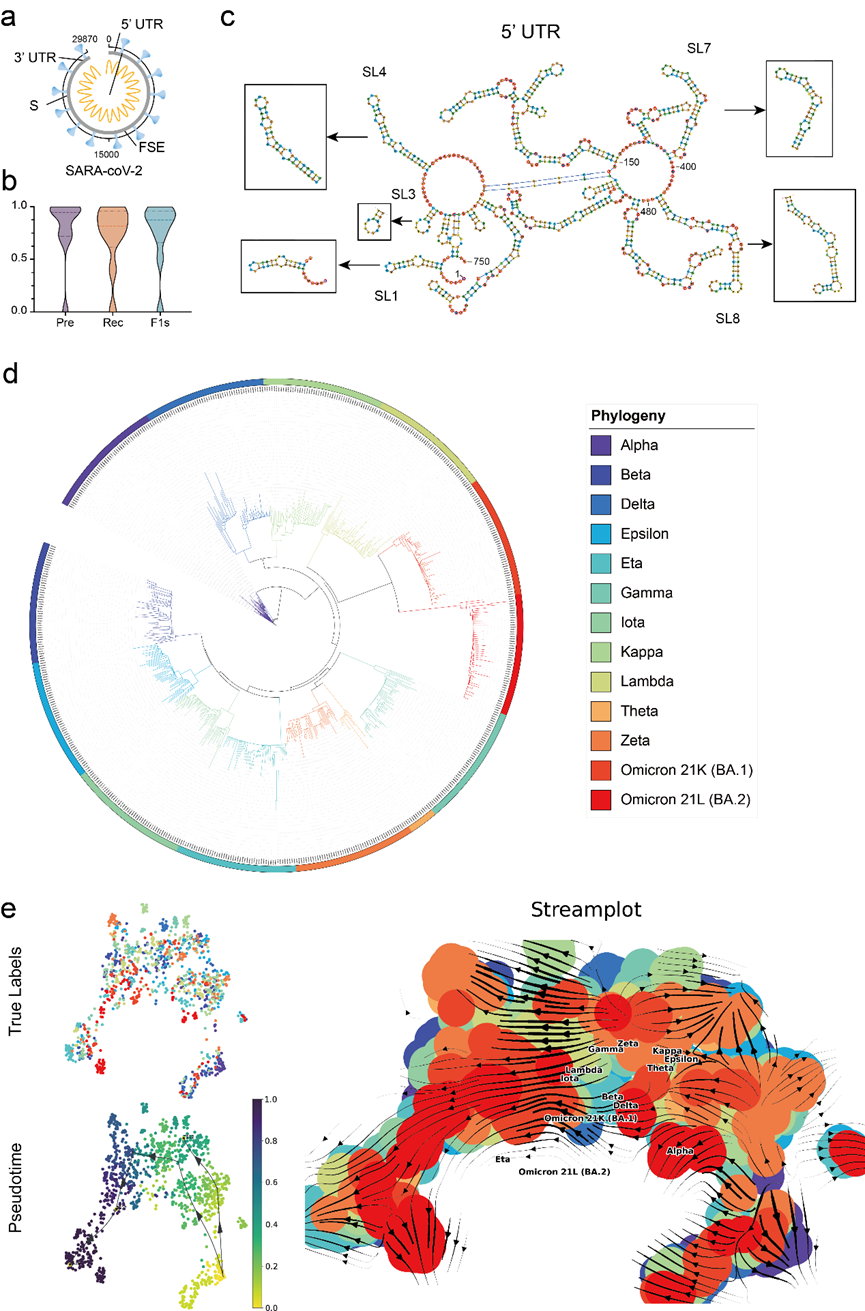
一般而言,分子的结构决定功能,RNA-FM 既然可以出色地完成 RNA 结构预测任务,那么是否能够利用 RNA-FM 也提升功能预测的结果呢?
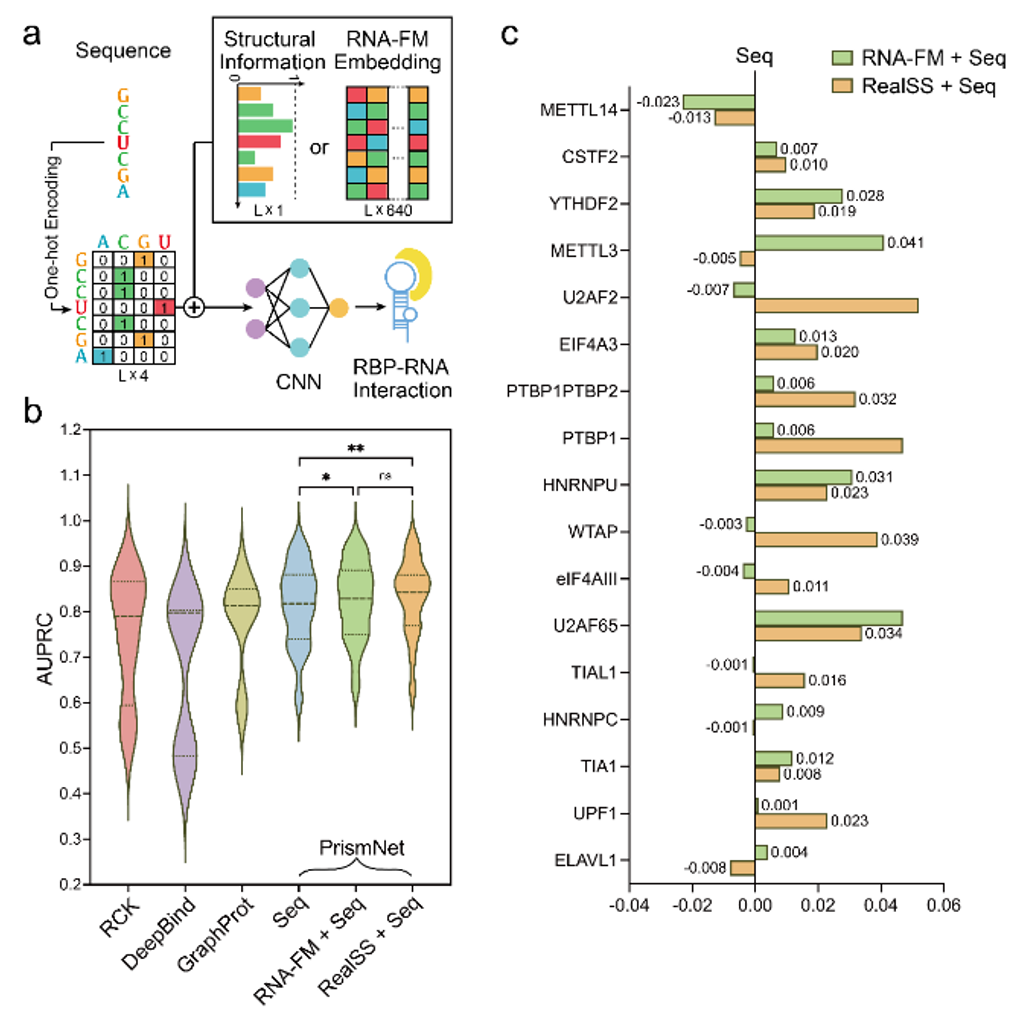
因此,文章进一步尝试将 RNA-FM 引入下游的 RNA 功能预测任务中,比如利用 RNA-FM 的 embedding 进行 RNA - 蛋白质作用的预测。
实验证明,RNA-FM embedding 的引入提升了模型的性能,且在一些例子中竟然达到了匹配真实二级结构信息作为输入的预测结果。
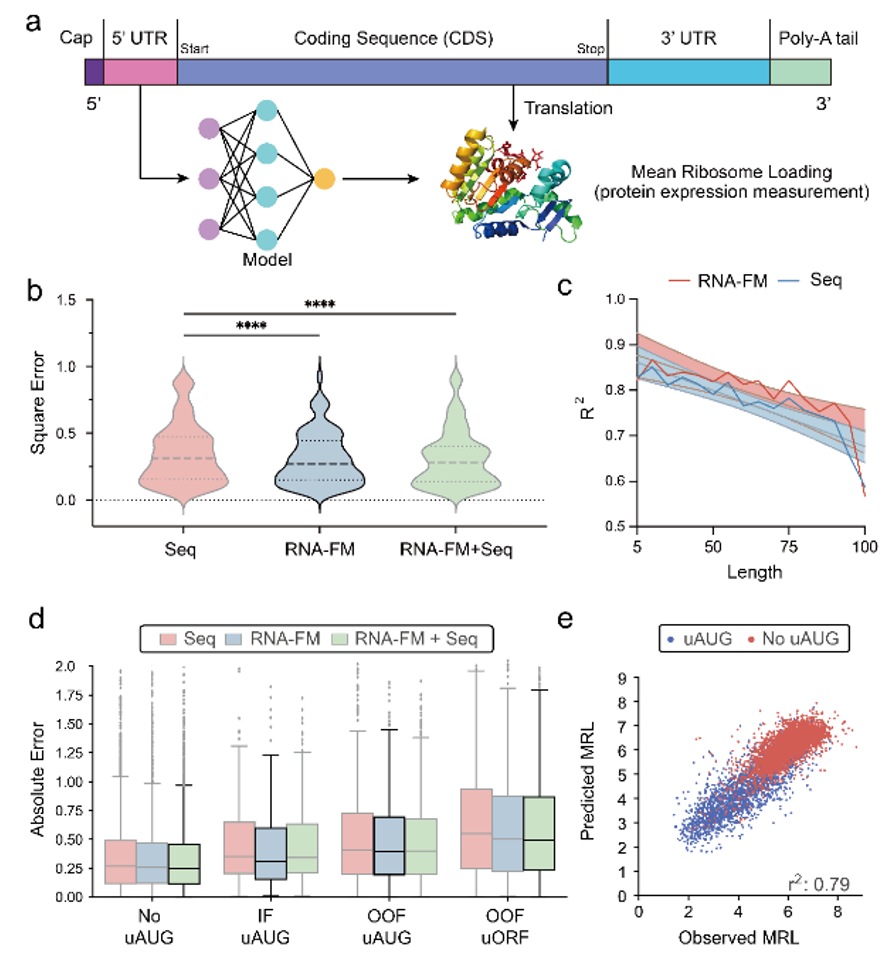
为了探究基于 ncRNA 训练的 RNA-FM 是否可以泛化到其他 RNA 上,文章最后尝试利用 RNA-FM 基于 mRNA 上的 5’UTR 进行蛋白表达的功能预测。虽然 mRNA 不属于 ncRNA,但是其上的 5‘UTR 是不翻译但具有调控功能的区域,符合 ncRNA 的特点,且未出现在训练数据中。
从下图可以看到,包含 RNA-FM embedding 的模型总是优于不包含的模型。尽管性能上的提升比较有限,但也部分说明 RNA-FM 在非 ncRNA 的数据上也具有一定的泛化性。
结论
总的来说,该文章以无标签的 RNA 序列数据预训练语言模型 RNA-FM,并通过直接或间接的方式,在结构或功能等一系列不同的任务上进行全面的验证,证明了 RNA-FM 确实可以有效地提升计算方法在下游任务中的表现。
RNA-FM 的出现一定程度上缓解了 RNA 带标注数据紧张的现状,为其他研究者提供了便捷的访问大批量的无标签数据的接口,其将以 RNA 领域基础模型的身份,为该领域的各种各样的研究提供强有力的支援与帮助。
作者简介
本文有两个共同第一作者。陈佳阳,香港中文大学研究助理。胡智航,香港中文大学在读博士生。
本文有两位通讯作者。孙思琦,复旦大学智能复杂体系实验室和上海人工智能实验室青年研究员,主页 https://intersun.github.io。
李煜,香港中文大学助理教授,MIT James Collins Lab 访问助理教授,Broad Institute of MIT and Harvard 研究科学家,哈佛大学 Wyss Institute 访问学者,Forbes 30 Under 30 Asia list–Class of 2022, Healthcare & Science。主页:https://liyu95.com。