介绍
嘿!几个小时前我刚刚完成一个深度学习项目,现在我想分享一下我所做的事情。这一挑战的目标是确定一个人是否患有肺炎。如果是,则确定是否由细菌或病毒引起。好吧,我觉得这个项目应该叫做分类而不是检测。
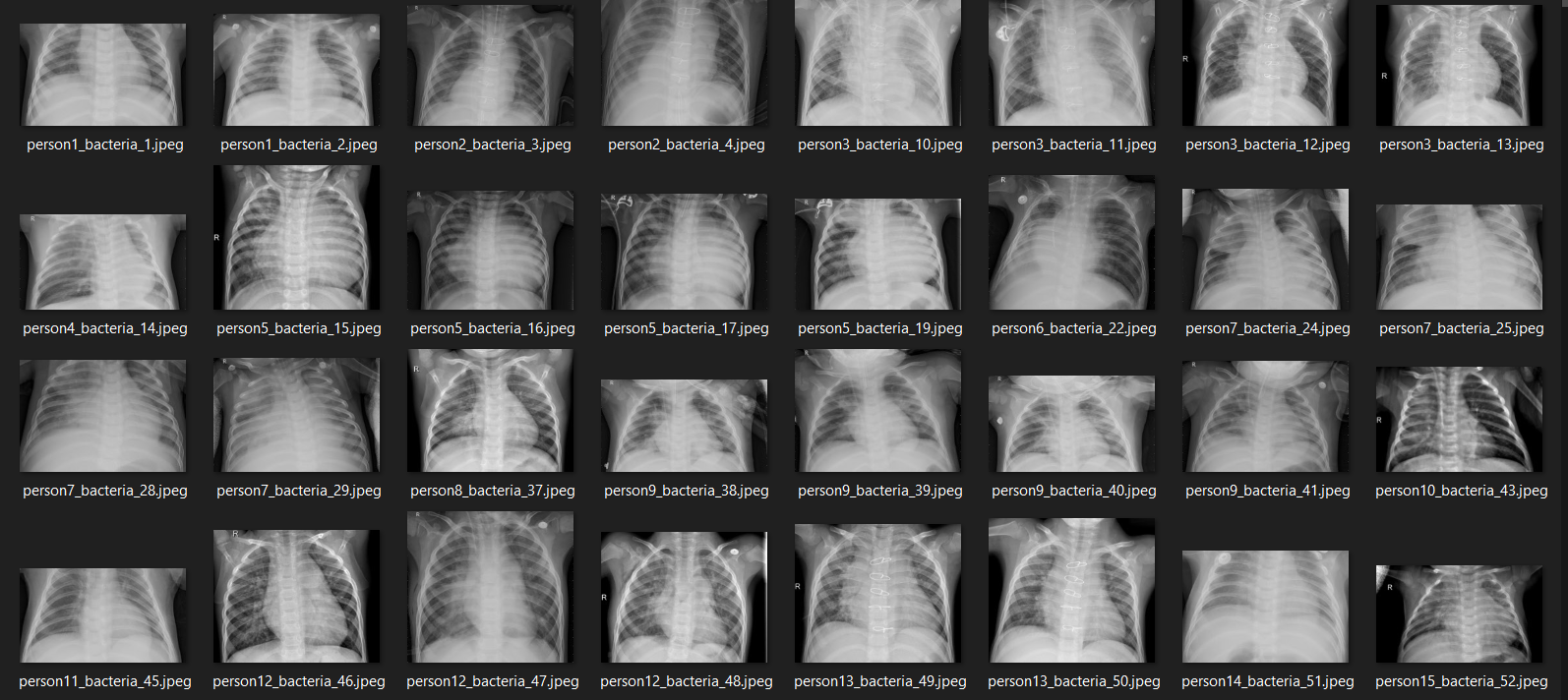
换句话说,此任务将是一个多分类问题,其中标签名称为:normal(正常),virus(病毒)和bacteria(细菌)。为了解决这个问题,我将使用CNN(卷积神经网络),它具有出色的图像分类能力,。不仅如此,在这里我还实现了图像增强技术,以提高模型性能。顺便说一句,我获得了80%的测试数据准确性,这对我来说是非常令人印象深刻的。
整个数据集本身的大小约为1 GB,因此下载可能需要一段时间。或者,我们也可以直接创建一个Kaggle Notebook并在那里编码整个项目,因此我们甚至不需要下载任何内容。接下来,如果浏览数据集文件夹,你将看到有3个子文件夹,即train,test和val。
好吧,我认为这些文件夹名称是不言自明的。此外,train文件夹中的数据分别包括正常,病毒和细菌类别的1341、1345和2530个样本。我想这就是我介绍的全部内容了,现在让我们进入代码的编写!
注意:我在本文结尾处放置了该项目中使用的全部代码。
加载模块和训练图像
使用计算机视觉项目时,要做的第一件事是加载所有必需的模块和图像数据本身。我使用tqdm模块显示进度条,稍后你将看到它有用的原因。
我最后导入的是来自Keras模块的ImageDataGenerator。该模块将帮助我们在训练过程中实施图像增强技术。
- import os
- import cv2import pickleimport numpy as np
- import matplotlib.pyplot as plt
- import seaborn as sns
- from tqdm import tqdm
- from sklearn.preprocessing import OneHotEncoder
- from sklearn.metrics import confusion_matrix
- from keras.models import Model, load_model
- from keras.layers import Dense, Input, Conv2D, MaxPool2D, Flatten
- from keras.preprocessing.image import ImageDataGeneratornp.random.seed(22)
接下来,我定义两个函数以从每个文件夹加载图像数据。乍一看,下面的两个功能可能看起来完全一样,但是在使用粗体显示的行上实际上存在一些差异。这样做是因为NORMAL和PNEUMONIA文件夹中的文件名结构略有不同。尽管有所不同,但两个功能执行的其他过程基本相同。
首先,将所有图像调整为200 x 200像素。
这一点很重要,因为所有文件夹中的图像都有不同的尺寸,而神经网络只能接受具有固定数组大小的数据。
接下来,基本上所有图像都存储有3个颜色通道,这对X射线图像来说是多余的。因此,我的想法是将这些彩色图像都转换为灰度图像。
- # Do not forget to include the last slash
- def load_normal(norm_path): norm_files = np.array(os.listdir(norm_path)) norm_labels = np.array(['normal']*len(norm_files))
- norm_images = [] for image in tqdm(norm_files):
- image = cv2.imread(norm_path + image) image = cv2.resize(image, dsize=(200,200))
- image = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY) norm_images.append(image)
- norm_images = np.array(norm_images) return norm_images, norm_labels
- def load_pneumonia(pneu_path): pneu_files = np.array(os.listdir(pneu_path)) pneu_labels = np.array([pneu_file.split('_')[1] for pneu_file in pneu_files])
- pneu_images = [] for image in tqdm(pneu_files):
- image = cv2.imread(pneu_path + image) image = cv2.resize(image, dsize=(200,200))
- image = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY) pneu_images.append(image)
- pneu_images = np.array(pneu_images) return pneu_images, pneu_labels
声明了以上两个函数后,现在我们可以使用它来加载训练数据了。如果你运行下面的代码,你还将看到为什么我选择在该项目中实现tqdm模块。
- norm_images, norm_labels = load_normal('/kaggle/input/chest-xray-pneumonia/chest_xray/train/NORMAL/')pneu_images, pneu_labels = load_pneumonia('/kaggle/input/chest-xray-pneumonia/chest_xray/train/PNEUMONIA/')
到目前为止,我们已经获得了几个数组:norm_images,norm_labels,pneu_images和pneu_labels。
带_images后缀的表示它包含预处理的图像,而带_labels后缀的数组表示它存储了所有基本信息(也称为标签)。换句话说,norm_images和pneu_images都将成为我们的X数据,其余的将成为y数据。
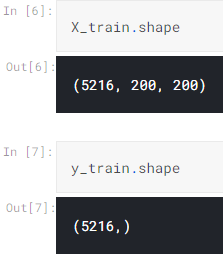
为了使项目看起来更简单,我将这些数组的值连接起来并存储在X_train和y_train数组中。
- X_train = np.append(norm_images, pneu_images, axis=0)
- y_train = np.append(norm_labels, pneu_labels)
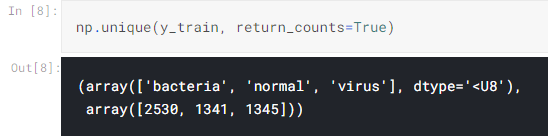
顺便说一句,我使用以下代码获取每个类的图像数:
显示多张图像
好吧,在这个阶段,显示几个图像并不是强制性的。但我想做是为了确保图片是否已经加载和预处理好。下面的代码用于显示14张从X_train阵列随机拍摄的图像以及标签。
- fig, axes = plt.subplots(ncols=7, nrows=2, figsize=(16, 4))
- indices = np.random.choice(len(X_train), 14)
- counter = 0
- for i in range(2):
- for j in range(7):
- axes[i,j].set_title(y_train[indices[counter]]) axes[i,j].imshow(X_train[indices[counter]], cmap='gray')
- axes[i,j].get_xaxis().set_visible(False) axes[i,j].get_yaxis().set_visible(False) counter += 1
- plt.show()
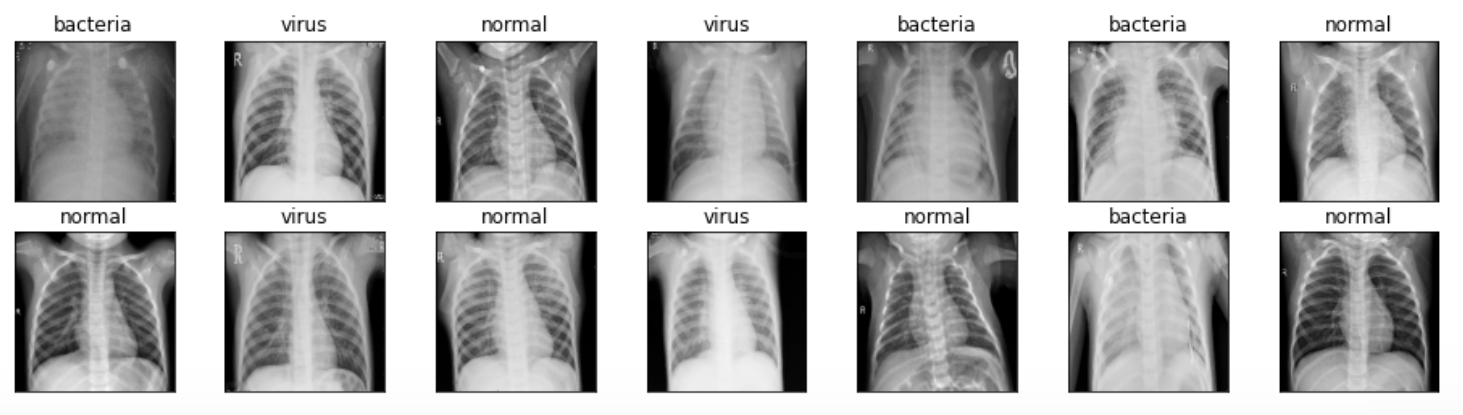
我们可以看到上图,所有图像现在都具有完全相同的大小,这与我用于本帖子封面图片的图像不同。
加载测试图像
我们已经知道所有训练数据都已成功加载,现在我们可以使用完全相同的函数加载测试数据。步骤几乎相同,但是这里我将那些加载的数据存储在X_test和y_test数组中。用于测试的数据本身包含624个样本。
- norm_images_test, norm_labels_test = load_normal('/kaggle/input/chest-xray-pneumonia/chest_xray/test/NORMAL/')pneu_images_test, pneu_labels_test = load_pneumonia('/kaggle/input/chest-xray-pneumonia/chest_xray/test/PNEUMONIA/')X_test = np.append(norm_images_test, pneu_images_test, axis=0)
- y_test = np.append(norm_labels_test, pneu_labels_test)
此外,我注意到仅加载整个数据集就需要很长时间。因此,我将使用pickle模块将X_train,X_test,y_train和y_test保存在单独的文件中。这样我下次想再使用这些数据的时候,就不需要再次运行这些代码了。
- # Use this to save variables
- with open('pneumonia_data.pickle', 'wb') as f:
- pickle.dump((X_train, X_test, y_train, y_test), f)# Use this to load variables
- with open('pneumonia_data.pickle', 'rb') as f:
- (X_train, X_test, y_train, y_test) = pickle.load(f)
由于所有X数据都经过了很好的预处理,因此现在使用标签y_train和y_test了。
标签预处理
此时,两个y变量都由以字符串数据类型编写的正常,细菌或病毒组成。实际上,这样的标签只是神经网络所不能接受的。因此,我们需要将其转换为单一格式。
幸运的是,我们从Scikit-Learn模块获取了 OneHotEncoder对象,它对完成转换非常有帮助。为此,我们需要先在y_train和y_test上创建一个新轴。(我们创建了这个新轴,因为那是OneHotEncoder期望的形状)。
- y_train = y_train[:, np.newaxis]
- y_test = y_test[:, np.newaxis]
接下来,像这样初始化one_hot_encoder。请注意,在这里我将False作为稀疏参数传递,以便简化下一步。但是,如果你想使用稀疏矩阵,则只需使用sparse = True或将参数保留为空即可。
- one_hot_encoder = OneHotEncoder(sparse=False)
最后,我们将使用one_hot_encoder将这些y数据转换为one-hot。然后将编码后的标签存储在y_train_one_hot和y_test_one_hot中。这两个数组是我们将用于训练的标签。
- y_train_one_hot = one_hot_encoder.fit_transform(y_train)
- y_test_one_hot = one_hot_encoder.transform(y_test)
将数据X重塑为(None,200,200,1)
现在让我们回到X_train和X_test。重要的是要知道这两个数组的形状分别为(5216、200、200)和(624、200、200)。
乍一看,这两个形状看起来还可以,因为我们可以使用plt.imshow()函数进行显示。但是,这种形状卷积层不可接受,因为它希望将一个颜色通道作为其输入。
因此,由于该图像本质上是灰度图像,因此我们需要添加一个1维的新轴,该轴将被卷积层识别为唯一的颜色通道。虽然它的实现并不像我的解释那么复杂:
- X_train = X_train.reshape(X_train.shape[0], X_train.shape[1], X_train.shape[2], 1)
- X_test = X_test.reshape(X_test.shape[0], X_test.shape[1], X_test.shape[2], 1)
运行上述代码后,如果我们同时检查X_train和X_test的形状,那么我们将看到现在的形状分别是(5216,200,200,1)和(624,200,200,1)。
数据扩充
增加数据(或者更具体地说是增加训练数据)的要点是,我们将通过创建更多的样本(每个样本都具有某种随机性)来增加用于训练的数据数量。这些随机性可能包括平移、旋转、缩放、剪切和翻转。
这种技术可以帮助我们的神经网络分类器减少过拟合,或者说,它可以使模型更好地泛化数据样本。幸运的是,由于存在可以从Keras模块导入的ImageDataGenerator对象,实现非常简单。
- datagen = ImageDataGenerator(
- rotation_range = 10,
- zoom_range = 0.1,
- width_shift_range = 0.1,
- height_shift_range = 0.1)
因此,我在上面的代码中所做的基本上是设置随机范围。
接下来,在初始化datagen对象之后,我们需要做的是使它和我们的X_train相匹配。然后,该过程被随后施加的flow()的方法,该步骤中是非常有用的,使得所述 train_gen对象现在能够产生增强数据的批次。
- datagen.fit(X_train)train_gen = datagen.flow(X_train, y_train_one_hot, batch_size=32)
CNN(卷积神经网络)
现在是时候真正构建神经网络架构了。让我们从输入层(input1)开始。因此,这一层基本上会获取X数据中的所有图像样本。因此,我们需要确保第一层接受与图像尺寸完全相同的形状。值得注意的是,我们仅需要定义(宽度,高度,通道),而不是(样本,宽度,高度,通道)。
此后,此输入层连接到几对卷积池层对,然后最终连接到全连接层。请注意,由于ReLU的计算速度比S型更快,因此模型中的所有隐藏层都使用ReLU激活函数,因此所需的训练时间更短。最后,要连接的最后一层是output1,它由3个具有softmax激活函数的神经元组成。
这里使用softmax是因为我们希望输出是每个类别的概率值。
- input1 = Input(shape=(X_train.shape[1], X_train.shape[2], 1))
- cnn = Conv2D(16, (3, 3), activation='relu', strides=(1, 1),
- padding='same')(input1)
- cnn = Conv2D(32, (3, 3), activation='relu', strides=(1, 1),
- padding='same')(cnn)
- cnn = MaxPool2D((2, 2))(cnn)
- cnn = Conv2D(16, (2, 2), activation='relu', strides=(1, 1),
- padding='same')(cnn)
- cnn = Conv2D(32, (2, 2), activation='relu', strides=(1, 1),
- padding='same')(cnn)
- cnn = MaxPool2D((2, 2))(cnn)
- cnn = Flatten()(cnn)cnn = Dense(100, activation='relu')(cnn)
- cnn = Dense(50, activation='relu')(cnn)
- output1 = Dense(3, activation='softmax')(cnn)
- model = Model(inputs=input1, outputs=output1)
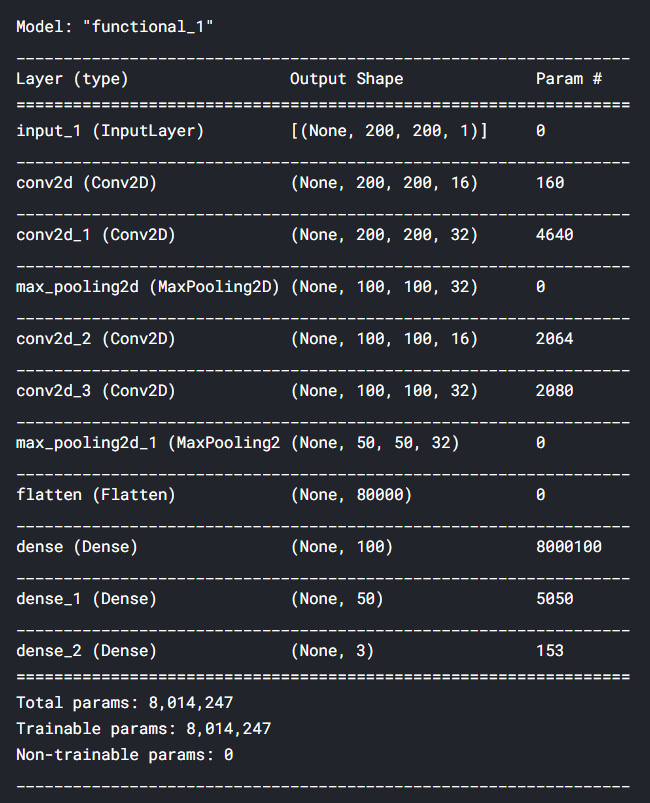
在使用上面的代码构造了神经网络之后,我们可以通过对model对象应用summary()来显示模型的摘要。下面是我们的CNN模型的详细情况。我们可以看到我们总共有800万个参数——这确实很多。好吧,这就是为什么我在Kaggle Notebook上运行这个代码。
总之,在构建模型之后,我们需要使用分类交叉熵损失函数和Adam优化器来编译神经网络。使用这个损失函数,因为它只是多类分类任务中常用的函数。同时,我选择Adam作为优化器,因为它是在大多数神经网络任务中最小化损失的最佳选择。
- model.compile(loss='categorical_crossentropy',
- optimizer='adam', metrics=['acc'])
现在是时候训练模型了!在这里,我们将使用fit_generator()而不是fit(),因为我们将从train_gen对象获取训练数据。如果你关注数据扩充部分,你会注意到train_gen是使用X_train和y_train_one_hot创建的。因此,我们不需要在fit_generator()方法中显式定义X-y对。
- history = model.fit_generator(train_gen, epochs=30,
- validation_data=(X_test, y_test_one_hot))
train_gen的特殊之处在于,训练过程中将使用具有一定随机性的样本来完成。因此,我们在X_train中拥有的所有训练数据都不会直接输入到神经网络中。取而代之的是,这些样本将被用作生成器的基础,通过一些随机变换生成一个新图像。
此外,该生成器在每个时期产生不同的图像,这对于我们的神经网络分类器更好地泛化测试集中的样本非常有利。下面是训练的过程。
- Epoch 1/30
- 163/163 [==============================] - 19s 114ms/step - loss: 5.7014 - acc: 0.6133 - val_loss: 0.7971 - val_acc: 0.7228
- .
- .
- .
- Epoch 10/30
- 163/163 [==============================] - 18s 111ms/step - loss: 0.5575 - acc: 0.7650 - val_loss: 0.8788 - val_acc: 0.7308
- .
- .
- .
- Epoch 20/30
- 163/163 [==============================] - 17s 102ms/step - loss: 0.5267 - acc: 0.7784 - val_loss: 0.6668 - val_acc: 0.7917
- .
- .
- .
- Epoch 30/30
- 163/163 [==============================] - 17s 104ms/step - loss: 0.4915 - acc: 0.7922 - val_loss: 0.7079 - val_acc: 0.8045
整个训练本身在我的Kaggle Notebook上花费了大约10分钟。所以要耐心点!经过训练后,我们可以绘制出准确度得分的提高和损失值的降低,如下所示:
- plt.figure(figsize=(8,6))
- plt.title('Accuracy scores')
- plt.plot(history.history['acc'])
- plt.plot(history.history['val_acc'])
- plt.legend(['acc', 'val_acc'])
- plt.show()plt.figure(figsize=(8,6))
- plt.title('Loss value')
- plt.plot(history.history['loss'])
- plt.plot(history.history['val_loss'])
- plt.legend(['loss', 'val_loss'])
- plt.show()
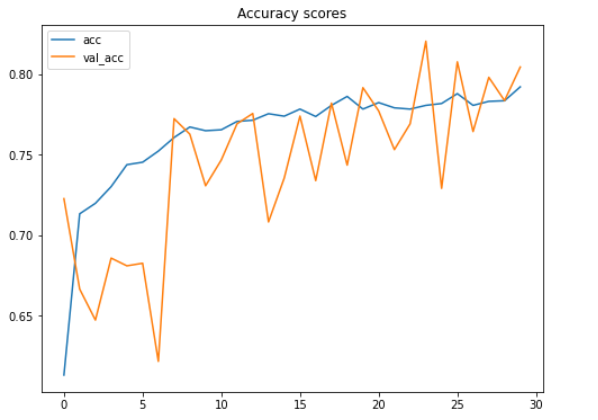
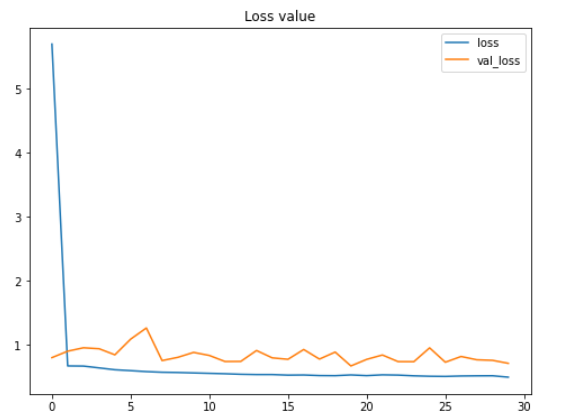
根据上面的两个图,我们可以说,即使在这30个时期内测试准确性和损失值都在波动,模型的性能仍在不断提高。
这里要注意的另一重要事情是,由于我们在项目的早期应用了数据增强方法,因此该模型不会遭受过拟合的困扰。我们在这里可以看到,在最终迭代中,训练和测试数据的准确性分别为79%和80%。
有趣的事实:在实施数据增强方法之前,我在训练数据上获得了100%的准确性,在测试数据上获得了64%的准确性,这显然是过拟合了。因此,我们可以在此处清楚地看到,增加训练数据对于提高测试准确性得分非常有效,同时也可以减少过拟合。
模型评估
现在,让我们深入了解使用混淆矩阵得出的测试数据的准确性。首先,我们需要预测所有X_test并将结果从独热格式转换回其实际的分类标签。
- predictions = model.predict(X_test)
- predictions = one_hot_encoder.inverse_transform(predictions)
接下来,我们可以像这样使用confusion_matrix()函数:
- cm = confusion_matrix(y_test, predictions)
重要的是要注意函数中使用的参数是(实际值,预测值)。该混淆矩阵函数的返回值是一个二维数组,用于存储预测分布。为了使矩阵更易于解释,我们可以使用Seaborn模块中的heatmap()函数进行显示。顺便说一句,这里的类名列表的值是根据one_hotencoder.categories返回的顺序获取的。
- classnames = ['bacteria', 'normal', 'virus']plt.figure(figsize=(8,8))
- plt.title('Confusion matrix')
- sns.heatmap(cm, cbar=False, xticklabels=classnames, yticklabels=classnames, fmt='d', annot=True, cmap=plt.cm.Blues)
- plt.xlabel('Predicted')
- plt.ylabel('Actual')
- plt.show()
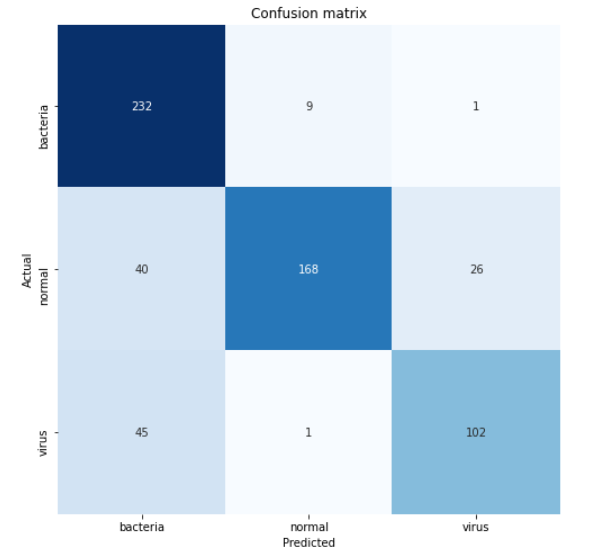
根据上面的混淆矩阵,我们可以看到45张病毒X射线图像被预测为细菌。这可能是因为很难区分这两种肺炎。但是,至少因为我们对242个样本中的232个进行了正确分类,所以我们的模型至少能够很好地预测由细菌引起的肺炎。
这就是整个项目!谢谢阅读!下面是运行整个项目所需的所有代码。
- import os
- import cv2import pickle # Used to save variablesimport numpy as npimport matplotlib.pyplot as pltimport seaborn as snsfrom tqdm import tqdm # Used to display progress bar
- from sklearn.preprocessing import OneHotEncoder
- from sklearn.metrics import confusion_matrix
- from keras.models import Model, load_model
- from keras.layers import Dense, Input, Conv2D, MaxPool2D, Flatten
- from keras.preprocessing.image import ImageDataGenerator # Used to generate images
- np.random.seed(22)
- # Do not forget to include the last slashdef load_normal(norm_path): norm_files = np.array(os.listdir(norm_path)) norm_labels = np.array(['normal']*len(norm_files))
- norm_images = [] for image in tqdm(norm_files):
- # Read image image = cv2.imread(norm_path + image) # Resize image to 200x200 px
- image = cv2.resize(image, dsize=(200,200))
- # Convert to grayscale image = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY) norm_images.append(image)
- norm_images = np.array(norm_images) return norm_images, norm_labels
- def load_pneumonia(pneu_path): pneu_files = np.array(os.listdir(pneu_path)) pneu_labels = np.array([pneu_file.split('_')[1] for pneu_file in pneu_files])
- pneu_images = [] for image in tqdm(pneu_files):
- # Read image image = cv2.imread(pneu_path + image) # Resize image to 200x200 px
- image = cv2.resize(image, dsize=(200,200))
- # Convert to grayscale image = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY) pneu_images.append(image)
- pneu_images = np.array(pneu_images) return pneu_images, pneu_labels
- print('Loading images')
- # All images are stored in _images, all labels are in _labelsnorm_images, norm_labels = load_normal('/kaggle/input/chest-xray-pneumonia/chest_xray/train/NORMAL/')
- pneu_images, pneu_labels = load_pneumonia('/kaggle/input/chest-xray-pneumonia/chest_xray/train/PNEUMONIA/')
- # Put all train images to X_train X_train = np.append(norm_images, pneu_images, axis=0)
- # Put all train labels to y_trainy_train = np.append(norm_labels, pneu_labels)
- print(X_train.shape)
- print(y_train.shape)
- # Finding out the number of samples of each classprint(np.unique(y_train, return_counts=True))print('Display several images')
- fig, axes = plt.subplots(ncols=7, nrows=2, figsize=(16, 4))
- indices = np.random.choice(len(X_train), 14)
- counter = 0
- for i in range(2):
- for j in range(7):
- axes[i,j].set_title(y_train[indices[counter]]) axes[i,j].imshow(X_train[indices[counter]], cmap='gray')
- axes[i,j].get_xaxis().set_visible(False) axes[i,j].get_yaxis().set_visible(False) counter += 1
- plt.show()print('Loading test images')
- # Do the exact same thing as what we have done on train datanorm_images_test, norm_labels_test = load_normal('/kaggle/input/chest-xray-pneumonia/chest_xray/test/NORMAL/')
- pneu_images_test, pneu_labels_test = load_pneumonia('/kaggle/input/chest-xray-pneumonia/chest_xray/test/PNEUMONIA/')
- X_test = np.append(norm_images_test, pneu_images_test, axis=0)
- y_test = np.append(norm_labels_test, pneu_labels_test)
- # Save the loaded images to pickle file for future use
- with open('pneumonia_data.pickle', 'wb') as f:
- pickle.dump((X_train, X_test, y_train, y_test), f)# Here's how to load it
- with open('pneumonia_data.pickle', 'rb') as f:
- (X_train, X_test, y_train, y_test) = pickle.load(f)
- print('Label preprocessing')
- # Create new axis on all y data
- y_train = y_train[:, np.newaxis]
- y_test = y_test[:, np.newaxis]
- # Initialize OneHotEncoder object
- one_hot_encoder = OneHotEncoder(sparse=False)
- # Convert all labels to one-hot
- y_train_one_hot = one_hot_encoder.fit_transform(y_train)
- y_test_one_hot = one_hot_encoder.transform(y_test)
- print('Reshaping X data')
- # Reshape the data into (no of samples, height, width, 1), where 1 represents a single color channel
- X_train = X_train.reshape(X_train.shape[0], X_train.shape[1], X_train.shape[2], 1)
- X_test = X_test.reshape(X_test.shape[0], X_test.shape[1], X_test.shape[2], 1)
- print('Data augmentation')
- # Generate new images with some randomness
- datagen = ImageDataGenerator(
- rotation_range = 10,
- zoom_range = 0.1,
- width_shift_range = 0.1,
- height_shift_range = 0.1)
- datagen.fit(X_train)
- train_gen = datagen.flow(X_train, y_train_one_hot, batch_size = 32)
- print('CNN')
- # Define the input shape of the neural network
- input_shape = (X_train.shape[1], X_train.shape[2], 1)
- print(input_shape)
- input1 = Input(shape=input_shape)
- cnn = Conv2D(16, (3, 3), activation='relu', strides=(1, 1),
- padding='same')(input1)
- cnn = Conv2D(32, (3, 3), activation='relu', strides=(1, 1),
- padding='same')(cnn)
- cnn = MaxPool2D((2, 2))(cnn)
- cnn = Conv2D(16, (2, 2), activation='relu', strides=(1, 1),
- padding='same')(cnn)
- cnn = Conv2D(32, (2, 2), activation='relu', strides=(1, 1),
- padding='same')(cnn)
- cnn = MaxPool2D((2, 2))(cnn)
- cnn = Flatten()(cnn)
- cnn = Dense(100, activation='relu')(cnn)
- cnn = Dense(50, activation='relu')(cnn)
- output1 = Dense(3, activation='softmax')(cnn)
- model = Model(inputs=input1, outputs=output1)
- model.compile(loss='categorical_crossentropy',
- optimizer='adam', metrics=['acc'])
- # Using fit_generator() instead of fit() because we are going to use data
- # taken from the generator. Note that the randomness is changing
- # on each epoch
- history = model.fit_generator(train_gen, epochs=30,
- validation_data=(X_test, y_test_one_hot))
- # Saving model
- model.save('pneumonia_cnn.h5')
- print('Displaying accuracy')
- plt.figure(figsize=(8,6))
- plt.title('Accuracy scores')
- plt.plot(history.history['acc'])
- plt.plot(history.history['val_acc'])
- plt.legend(['acc', 'val_acc'])
- plt.show()
- print('Displaying loss')
- plt.figure(figsize=(8,6))
- plt.title('Loss value')
- plt.plot(history.history['loss'])
- plt.plot(history.history['val_loss'])
- plt.legend(['loss', 'val_loss'])
- plt.show()
- # Predicting test data
- predictions = model.predict(X_test)
- print(predictions)
- predictions = one_hot_encoder.inverse_transform(predictions)
- print('Model evaluation')
- print(one_hot_encoder.categories_)
- classnames = ['bacteria', 'normal', 'virus']
- # Display confusion matrix
- cm = confusion_matrix(y_test, predictions)
- plt.figure(figsize=(8,8))
- plt.title('Confusion matrix')
- sns.heatmap(cm, cbar=False, xticklabels=classnames, yticklabels=classnames, fmt='d', annot=True, cmap=plt.cm.Blues)
- plt.xlabel('Predicted')
- plt.ylabel('Actual')
- plt.show()