本文转自雷锋网,如需转载请至雷锋网官网申请授权。
对于机器学习而言,区分异常数据或有显著差异数据至关重要。谷歌在 NeurIPS 2019 论文中提出并发布了针对基因组序列 OOD 检测的现实基准数据集,进而提出一种基于似然比的解决方案,可显著提高 OOD 检测的准确性。AI 科技评论将谷歌对该方法的官方解读编译如下。
深度学习科学家要成功部署机器学习系统,需要系统能够区分出异常数据或与训练中使用的数据有显着差异的数据。
由于深度神经网络分类器可能会将以高置信度将分布外(ODD)的输入分类到分布内的类别中,因此区分异常数据或有显著差异数据是十分重要的。当我们利用这些预测为现实决策提供依据时,异常数据检测将尤为重要。
例如,将机器学习模型应用于基于基因组序列的细菌检测,就是一项具有挑战性的现实应用。细菌检测对于败血症等传染病的诊断和治疗,以及食源性病原体的鉴定都非常关键。
近些年来,随着新细菌种类不断被发现,虽然基于已知分类训练的神经网络分类器通过交叉验证达到了很高的测量准确性,但部署模型仍具有高的挑战性,因为现实数据在不断进化发展,并且将不可避免地包含以往训练数据中从未出现过的基因组(OOD 输入)。
图1 近些年来,随着新的细菌种类逐渐地被发现。将已知的分类数据输入基于已知分类数据训练的分类器能够达到很高的准确性,这是因为输入的分类数据是已知的,但它可能将已知分类数据中混合了未知分类数据(如:ODD 数据)的输入进行错误的分类,并且具有很高的置信度。
在 NeurIPS 2019 发表的论文《分布外检测的似然比》(Likelihood Ratios for Out-of-Distribution Detection)中,谷歌受到上述新细菌种类检测这类现实问题的启发,提出并发布了针对基因组序列 OOD 检测的现实基准数据集。
-
论文地址:https://arxiv.org/abs/1906.02845
-
实现代码 GitHub 地址:https://github.com/google-research/google-research/tree/master/genomics_ood
他们利用基于基因组序列的生成模型测试了 OOD 检测的现有方法,发现似然值——即输入来自使用分布不均数据进行估算的分布数据的模型概率——通常是错误的。在最近的图像深度生成模型研究工作中,他们也观察到了这种现象,并通过统计背景影响来解释这种现象,进而提出一种基于似然比的解决方案,可以显著地提高 OOD 检测的准确性。
一、为什么密度模型无法应用于 OOD 检测?
为了模拟实际问题并系统地评估不同的方法,他们建立了一个新的细菌数据集,使用的数据来自美国国家生物技术信息中心(NCBI )对外开放的原核生物目录基因组序列数据。
为了模拟测序数据,他们将基因组片段化为当前测序技术通常使用的 250 个短序列长度的碱基对。然后,其将分布内和分布外的数据按发现日期进行分离,以便截止时间之前被发现的细菌种类被定义在分布内,在之后发现的被定义为分布外(OOD) 。
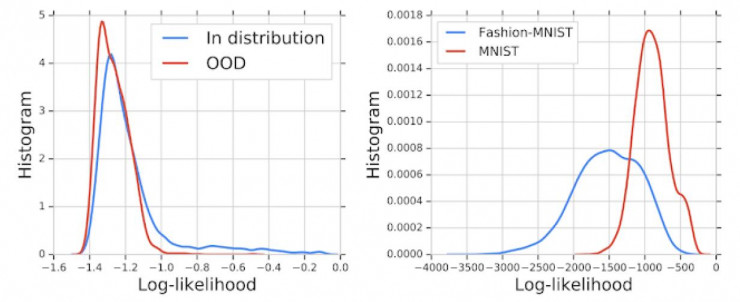
然后,他们基于分布内的基因组序列训练深度生成模型,通过绘制似然值曲线,检验模型辨别输入的分布内和分布外数据的能力。 OOD 序列似然值的直方图与分布内序列似然值高度重合,则表明生成模型无法区分在两个种类之间进行的 OOD 检测结果。
在图像深度生成模型的早期研究中(相关阅读参考:https://arxiv.org/abs/1810.09136)也得到了类似的结论。例如,利用 Fashion-MNIST 数据集(由衣服和鞋类的图像组成)训练 PixelCNN ++ 模型,比来自 MNIST 数据集(包括数字0-9的图像)的 OOD 图像分配了更高的似然值。
图2 左:分布内和分布外(OOD)基因组序列的似然值直方图。 似然值未能分辨出分布内和OOD基因组序列。 右:Fashion-MNIST 数据集训练模型、MNIST 数据集估计的似然值直方图。 模型在OOD(MNIST)图像上比在分布内图像分配了更高的似然值。
在研究这种失败模型时,他们观察到背景统计可能影响了似然值的计算。为了更直观地理解该现象,假设输入由两个部分组成:(1)以背景统计为特征的背景成分,(2)以指定于分布内数据专用的模式为特征的语义成分。
例如,可以将 MNIST 图像建模为背景加语义。当人类解读图像时,可以轻松地忽略背景信息而主要关注语义信息,例如下图中的“ /”标记。但是当为图像中的所有像素计算似然值时,计算结果中同时包括了语义像素和背景像素。虽然他们只需使用语义的似然值进行决策,但原始的似然值结果中可能大多数都是背景成分。
图3 左上:Fashion-MNIST 的示例图像。 左下:MNIST 的示例图像。 右:MNIST 图像中的背景和语义成分。
二、OOD 检测的似然比
他们提出了一种去除背景影响并专注于语义成分的似然比方法。
首先,受遗传突变的启发,他们利用扰动输入方法训练背景模型,并通过随机选择输入值的位置,将其替换为另一个具有相等概率的值。为了成像,他们从从256个可能的像素值中随机选择输入值;针对DNA 序列,他们从四个可能的核苷酸(A,T,C或G)中选出输入值。此过程中,适量的扰动会破坏数据的语义结构,导致只能捕获到背景。
接着,他们计算完整模型与背景模型之间的似然比,去掉了背景成分,这样就只保留了语义的似然值。似然比是背景对比得分,即它抓住了语义与背景对比的意义。
为了定性评估似然值与似然比之间的差异,他们绘制了在 Fashion-MNIST 数据集和 MNIST 数据集中每个像素的似然值和似然比值,创建了与图像相同的尺寸的热图。
这使他们可以分别直观地看到哪些像素对于这两项值的贡献最大。从对数似然热图中可以看到,对于似然值而言,背景像素比语义像素的贡献更多。
事后看来这并不足为奇,这是由于背景像素主要由一连串零组成,因此很容易被模型学习。
MNIST 和 Fashion-MNIST 热图之间的比较则说明了为什么 MNIST 返回更高的似然值——仅仅是因为它包含了更多的背景像素!相反,似然比的结果更多地集中在语义像素上。
这种似然比方法修正了背景影响。他们基于 Fashion-MNIST 训练 PixelCNN ++ 模型,然后在 MNIST 图像数据集进行 OOD 检测,实验结果得到了显著改善,AUROC 评分从 0.089 提高至 0.994 。
当他们将似然比方法应用于基因组基准数据集这一极具挑战的问题时,对比其它 12 种基线方法,该方法表现出了最佳性能。
不过他们也表示,尽管该似然比方法在基因组数据集上达到了最先进的性能,但离将模型部署到实际应用中的高准确性要求仍存在一定距离。 他们鼓励研究人员努力去解决这一重要问题,并改善当前的最新技术。