一、通过重新构建数据进行整形
数据整形最直接的思路就把数据全部向量化,然后按要求用向量构建其他类型的数据。这样是不是会产生大量的中间变量、占用大量内存?没错。R语言的任何函数(包括赋值)操作都会有同样的问题,因为R函数的参数传递方式是传值不传址,变量不可能原地址修改后再放回原地址。
矩阵和多维数组的向量化有直接的类型转换函数: as.vector,向量化后的结果顺序是先列后行再其他:
- > (x <- matrix(1:4, ncol=2)) #为节省空间,下面的结果省略了一些空行
- [,1] [,2]
- [1,] 1 3
- [2,] 2 4
- > as.vector(x)
- [1] 1 2 3 4
- > (x <- array(1:8, dim=c(2,2,2)))
- , , 1
- [,1] [,2]
- [1,] 1 3
- [2,] 2 4
- , , 2
- [,1] [,2]
- [1,] 5 7
- [2,] 6 8
- > as.vector(x)
- [1] 1 2 3 4 5 6 7 8
列表向量化可以用unlist,数据框本质是元素长度相同的列表,所以也用unlist:
- > (x <- list(x=1:3, y=5:10))
- $x
- [1] 1 2 3
- $y
- [1] 5 6 7 8 9 10
- > unlist(x)
- x1 x2 x3 y1 y2 y3 y4 y5 y6
- 1 2 3 5 6 7 8 9 10
- > x <- data.frame(x=1:3, y=5:7)
- > unlist(x)
- x1 x2 x3 y1 y2 y3
- 1 2 3 5 6 7
其他类型的数据一般都可以通过数组、矩阵或列表转成向量。一些软件包有自定义的数据类型,如果考虑周到的话应该会有合适的类型转换函数。
二、transform 和 within函数
transform 函数对数据框进行操作,作用是为原数据框增加新的列变量。但应该注意的是“原数据框”根本不是原来的那个数据框,而是一个它的拷贝。下面代码为airquality数据框增加了一列log.ozone,但因为没有把结果赋值给原变量名,所以原数据是不变的:
- > head(airquality,2)
- Ozone Solar.R Wind Temp Month Day
- 1 41 190 7.4 67 5 1
- 2 36 118 8.0 72 5 2
- > aq <- transform(airquality, loglog.ozone=log(Ozone))
- > head(airquality,2)
- Ozone Solar.R Wind Temp Month Day
- 1 41 190 7.4 67 5 1
- 2 36 118 8.0 72 5 2
- > head(aq,2)
- Ozone Solar.R Wind Temp Month Day log.ozone
- 1 41 190 7.4 67 5 1 3.713572
- 2 36 118 8.0 72 5 2 3.583519
transform可以增加新列变量,可以改变列变量的值,也可以通过NULL赋值的方式删除列变量:
- > aq <- transform(airquality, loglog.ozone=log(Ozone), Ozone=NULL, WindWind=Wind^2)
- > head(aq,2)
- Solar.R Wind Temp Month Day log.ozone
- 1 190 54.76 67 5 1 3.713572
- 2 118 64.00 72 5 2 3.583519
- > aq <- transform(airquality, loglog.ozone=log(Ozone), Ozone=NULL, Month=NULL, WindWind=Wind^2)
- > head(aq,2)
- Solar.R Wind Temp Day log.ozone
- 1 190 54.76 67 1 3.713572
- 2 118 64.00 72 2 3.583519
within 比 transform 灵活些,除数据框外还可以使用其他类型数据,但用法不大一样,而且函数似乎也不够完善:
- > aq <- within(airquality, {
- + log.ozone <- log(Ozone)
- + squared.wind <- Wind^2
- + rm(Ozone, Wind)
- + } )
- > head(aq,2)
- Solar.R Temp Month Day squared.wind log.ozone
- 1 190 67 5 1 54.76 3.713572
- 2 118 72 5 2 64.00 3.583519
- > (x <- list(a=1:3, b=letters[3:10], c=LETTERS[9:14]))
- $a
- [1] 1 2 3
- $b
- [1] "c" "d" "e" "f" "g" "h" "i" "j"
- $c
- [1] "I" "J" "K" "L" "M" "N"
- > within(x, {log.a <- log(a); d <- paste(b, c, sep=':'); rm(b)})
- $a
- [1] 1 2 3
- $c
- [1] "I" "J" "K" "L" "M" "N"
- $d
- [1] "c:I" "d:J" "e:K" "f:L" "g:M" "h:N" "i:I" "j:J"
- $log.a
- [1] 0.0000000 0.6931472 1.0986123
- > within(x, {log.a <- log(a); d <- paste(b, c, sep=':'); rm(b,c)})
- $a
- [1] 1 2 3
- $b #为什么删除两个列表元素会得到这样的结果?
- NULL
- $c
- NULL
- $d
- [1] "c:I" "d:J" "e:K" "f:L" "g:M" "h:N" "i:I" "j:J"
- $log.a
- [1] 0.0000000 0.6931472 1.0986123
三、reshape、stack和unstack 函数
reshape是R base/stats的函数,主要用于数据框长格式和宽格式之间的转换。reshape函数的参数很多,不容易记,牛人Hadley Wickham搞出reshape和reshape2包以后这个函数几乎被人遗忘:
- reshape(data, varying = NULL, v.names = NULL, timevar = "time",
- idvar = "id", ids = 1:NROW(data),
- times = seq_along(varying[[1]]),
- drop = NULL, direction, new.row.names = NULL,
- sep = ".",
- split = if (sep == "") {
- list(regexp = "[A-Za-z][0-9]", include = TRUE)
- } else {
- list(regexp = sep, include = FALSE, fixed = TRUE)}
- )
既然可以被遗忘,那就等你走投无路的时候(估计不会有这样的情况发生)再去了解它吧。
stack 和 unstack 的作用和reshape类似,用于数据框/列表的长、宽格式之间转换。数据框宽格式是我们记录原始数据常用的格式,类似这样:
- > x <- data.frame(CK=c(1.1, 1.2, 1.1, 1.5), T1=c(2.1, 2.2, 2.3, 2.1), T2=c(2.5, 2.2, 2.3, 2.1))
- > x
- CK T1 T2
- 1 1.1 2.1 2.5
- 2 1.2 2.2 2.2
- 3 1.1 2.3 2.3
- 4 1.5 2.1 2.1
一般统计和作图用的是长格式,stack可以做这个:
- > (xx <- stack(x))
- values ind
- 1 1.1 CK
- 2 1.2 CK
- 3 1.1 CK
- 4 1.5 CK
- 5 2.1 T1
- 6 2.2 T1
- 7 2.3 T1
- 8 2.1 T1
- 9 2.5 T2
- 10 2.2 T2
- 11 2.3 T2
- 12 2.1 T2
而unstack的作用正好和stack相反,但是要注意它的第二个参数是公式类型:公式左边的变量是值,右边的变量会被当成因子类型,它的每个水平都会形成一列:
- > unstack(xx, values~ind)
- CK T1 T2
- 1 1.1 2.1 2.5
- 2 1.2 2.2 2.2
- 3 1.1 2.3 2.3
- 4 1.5 2.1 2.1
四、reshape/reshape2 包
Hadley Wickham,牛人,很牛X的一个人,写了很多R语言包,著名的有ggplot2, plyr, reshape/reshape2等。reshape2包是reshape包的重写版,用reshape2就行,都在CRAN源中,用install.packages函数就可以安装。reshape/reshape2的函数很少,一般用户直接使用的是melt, acast 和 dcast 函数。
melt是溶解/分解的意思,即拆分数据。reshape/reshape2的melt函数是个S3通用函数,它会根据数据类型(数据框,数组或列表)选择melt.data.frame, melt.array 或 melt.list函数进行实际操作。
如果是数组(array)类型,melt的用法就很简单,它依次对各维度的名称进行组合将数据进行线性/向量化。如果数组有n维,那么得到的结果共有n+1列,前n列记录数组的位置信息,最后一列才是观测值:
- > datax <- array(1:8, dim=c(2,2,2))
- > melt(datax)
- Var1 Var2 Var3 value
- 1 1 1 1 1
- 2 2 1 1 2
- 3 1 2 1 3
- 4 2 2 1 4
- 5 1 1 2 5
- 6 2 1 2 6
- 7 1 2 2 7
- 8 2 2 2 8
- > melt(datax, varnames=LETTERS[24:26],value.name="Val")
- X Y Z Val
- 1 1 1 1 1
- 2 2 1 1 2
- 3 1 2 1 3
- 4 2 2 1 4
- 5 1 1 2 5
- 6 2 1 2 6
- 7 1 2 2 7
- 8 2 2 2 8
如果是列表数据,melt 函数将列表中的数据拉成两列,一列记录列表元素的值,另一列记录列表元素的名称;如果列表中的元素是列表,则增加列变量存储元素名称。元素值排列在前,名称在后,越是顶级的列表元素名称越靠后:
- > datax <- list(agi="AT1G10000", GO=c("GO:1010","GO:2020"), KEGG=c("0100", "0200", "0300"))
- > melt(datax)
- value L1
- 1 AT1G10000 agi
- 2 GO:1010 GO
- 3 GO:2020 GO
- 4 0100 KEGG
- 5 0200 KEGG
- 6 0300 KEGG
- > melt(list(at_0100=datax))
- value L2 L1
- 1 AT1G10000 agi at_0100
- 2 GO:1010 GO at_0100
- 3 GO:2020 GO at_0100
- 4 0100 KEGG at_0100
- 5 0200 KEGG at_0100
- 6 0300 KEGG at_0100
如果数据是数据框类型,melt的参数就稍微复杂些:
- melt(data, id.vars, measure.vars,
- variable.name = "variable", ..., na.rm = FALSE,
- value.name = "value")
其中 id.vars 是被当做维度的列变量,每个变量在结果中占一列;measure.vars 是被当成观测值的列变量,它们的列变量名称和值分别组成 variable 和 value两列,列变量名称用variable.name 和 value.name来指定。我们用airquality数据来看看:
- > str(airquality)
- 'data.frame': 153 obs. of 6 variables:
- $ Ozone : int 41 36 12 18 NA 28 23 19 8 NA ...
- $ Solar.R: int 190 118 149 313 NA NA 299 99 19 194 ...
- $ Wind : num 7.4 8 12.6 11.5 14.3 14.9 8.6 13.8 20.1 8.6 ...
- $ Temp : int 67 72 74 62 56 66 65 59 61 69 ...
- $ Month : int 5 5 5 5 5 5 5 5 5 5 ...
- $ Day : int 1 2 3 4 5 6 7 8 9 10 ...
如果打算按月份分析臭氧和太阳辐射、风速、温度三者(列2:4)的关系,我们把它转成长格式数据框:
- > aq <- melt(airquality, var.ids=c("Ozone", "Month", "Day"),
- + measure.vars=c(2:4), variable.name="V.type", value.name="value")
- > str(aq)
- 'data.frame': 459 obs. of 5 variables:
- $ Ozone : int 41 36 12 18 NA 28 23 19 8 NA ...
- $ Month : int 5 5 5 5 5 5 5 5 5 5 ...
- $ Day : int 1 2 3 4 5 6 7 8 9 10 ...
- $ V.type: Factor w/ 3 levels "Solar.R","Wind",..: 1 1 1 1 1 1 1 1 1 1 ...
- $ value : num 190 118 149 313 NA NA 299 99 19 194 ...
var.ids 可以写成id,measure.vars可以写成measure。id(即var.ids)和 观测值(即measure.vars)这两个参数可以只指定其中一个,剩余的列被当成另外一个参数的值;如果两个都省略,数值型的列被看成观测值,其他的被当成id。如果想省略参数或者去掉部分数据,参数名最好用 id/measure,否则得到的结果很可能不是你要的:
- > str(melt(airquality, var.ids=c(1,5,6), measure.vars=c(2:4)))
- 'data.frame': 459 obs. of 5 variables:
- $ Ozone : int 41 36 12 18 NA 28 23 19 8 NA ...
- $ Month : int 5 5 5 5 5 5 5 5 5 5 ...
- $ Day : int 1 2 3 4 5 6 7 8 9 10 ...
- $ variable: Factor w/ 3 levels "Solar.R","Wind",..: 1 1 1 1 1 1 1 1 1 1 ...
- $ value : num 190 118 149 313 NA NA 299 99 19 194 ...
- > str(melt(airquality, var.ids=1, measure.vars=c(2:4))) #看这里,虽然id只引用了一列,但结果却不是这样
- 'data.frame': 459 obs. of 5 variables:
- $ Ozone : int 41 36 12 18 NA 28 23 19 8 NA ...
- $ Month : int 5 5 5 5 5 5 5 5 5 5 ...
- $ Day : int 1 2 3 4 5 6 7 8 9 10 ...
- $ variable: Factor w/ 3 levels "Solar.R","Wind",..: 1 1 1 1 1 1 1 1 1 1 ...
- $ value : num 190 118 149 313 NA NA 299 99 19 194 ...
- > str(melt(airquality, var.ids=1)) #这样用更惨,结果不是我们要的吧?
- Using as id variables
- 'data.frame': 918 obs. of 2 variables:
- $ variable: Factor w/ 6 levels "Ozone","Solar.R",..: 1 1 1 1 1 1 1 1 1 1 ...
- $ value : num 41 36 12 18 NA 28 23 19 8 NA ...
- > str(melt(airquality, id=1)) #这样才行
- 'data.frame': 765 obs. of 3 variables:
- $ Ozone : int 41 36 12 18 NA 28 23 19 8 NA ...
- $ variable: Factor w/ 5 levels "Solar.R","Wind",..: 1 1 1 1 1 1 1 1 1 1 ...
- $ value : num 190 118 149 313 NA NA 299 99 19 194 ...
数据整容有什么用?当然有。别忘了reshape2和ggplot2都是Hadley Wickham的作品,melt 以后的数据(称为molten数据)用ggplot2做统计图就很方便了,可以快速做出我们需要的图形:
- library(ggplot2)
- aq$Month <- factor(aq$Month)
- p <- ggplot(data=aq, aes(x=Ozone, y=value, color=Month)) + theme_bw()
- p + geom_point(shape=20, size=4) + geom_smooth(aes(group=1), fill="gray80") + facet_wrap(~V.type, scales="free_y")
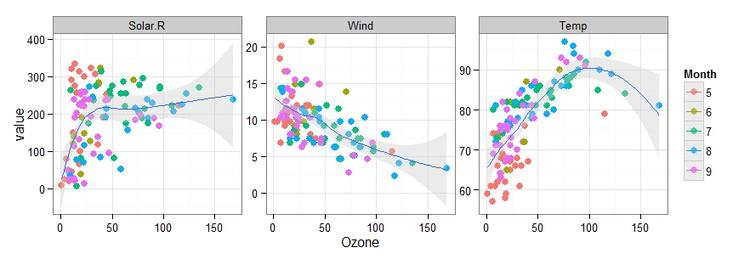
melt获得的数据(molten data)可以用 acast 或 dcast 还原。acast获得数组,dcast获得数据框。和unstack函数一样,cast函数使用公式参数。公式的左边每个变量都会作为结果中的一列,而右边的变量被当成因子类型,每个水平都会在结果中产生一列。
- > head(dcast(aq, Ozone+Month+Day~V.type))
- Ozone Month Day Solar.R Wind Temp
- 1 1 5 21 8 9.7 59
- 2 4 5 23 25 9.7 61
- 3 6 5 18 78 18.4 57
- 4 7 5 11 NA 6.9 74
- 5 7 7 15 48 14.3 80
- 6 7 9 24 49 10.3 69
cast函数的作用不只是还原数据,还可以使用函数对数据进行汇总(aggregate)。事实上,melt函数是为cast服务的,目的是使用cast函数对数据进行aggregate:
- > dcast(aq, Month~V.type, fun.aggregate=mean, na.rm=TRUE)
- Month Solar.R Wind Temp
- 1 5 181.2963 11.622581 65.54839
- 2 6 190.1667 10.266667 79.10000
- 3 7 216.4839 8.941935 83.90323
- 4 8 171.8571 8.793548 83.96774
- 5 9 167.4333 10.180000 76.90000
五、plyr 包
plyr 的功能已经远远超出数据整容的范围,Hadley在plyr中应用了split-apply-combine的数据处理哲学,即:先将数据分离,然后应用某些处理函数,最后将结果重新组合成所需的形式返回。某些人士喜欢用“揉”来表述这样的数据处理;“揉”,把数据当面团捣来捣去,很哲,砖家们的砖头落下来,拍死人绝不偿命。
先别哲了,来点实际的:plyr的函数命名方式比较规律,很容易记忆和使用。比如 a开头的函数aaply, adply 和 alply 将数组(array)分别转成数组、数据框和列表;daply, ddply 和 dlply 将数据框分别转成数组、数据框和列表;而laply, ldaply, llply将列表(list)分别转成数组、数据框和列表。
下面我们看看如何使用ldply函数将ath1121501.db包中的KEGG列表数据转成数据框:
- > library(ath1121501.db)
- > keggs <- as.list(ath1121501PATH[mappedkeys(ath1121501PATH)])
- > head(ldply(keggs, paste, collapse='; '))
- .id V1
- 1 261579_at 00190
- 2 261569_at 04712
- 3 261583_at 00010; 00020; 00290; 00620; 00650; 01100; 01110
- 4 261574_at 00903; 00945; 01100; 01110
- 5 261043_at 00051; 00520; 01100
- 6 261044_at 04122
plyr包的函数较多,不再一一介绍,更多用法请参考它的在线帮助,Hadley 也写了很详细的tutorial:http://plyr.had.co.nz/09-user/
原文链接:http://helloxxxxxx.blog.163.com/blog/static/21601509520133343821837/?latestBlog
【编辑推荐】