Alphafold可能成为第一个获诺贝尔奖的深度学习模型? 原创 精华
诺贝尔奖主要颁给那些为人类做出巨大贡献的人,我国的屠呦呦就因发现青蒿素在2015年获得诺贝尔医学奖。
Alphafold不是某个人类科学家,它是Google DeepMind开发的一个深度学习模型,它能够预测蛋白质结构。
把诺奖颁给一个模型,你可能会觉得我危言耸听了,但如果你了解蛋白质结构的作用,你肯定会改变想法了。
一、蛋白质合成过程
在此之前还是先简单了解下蛋白质的合成过程。
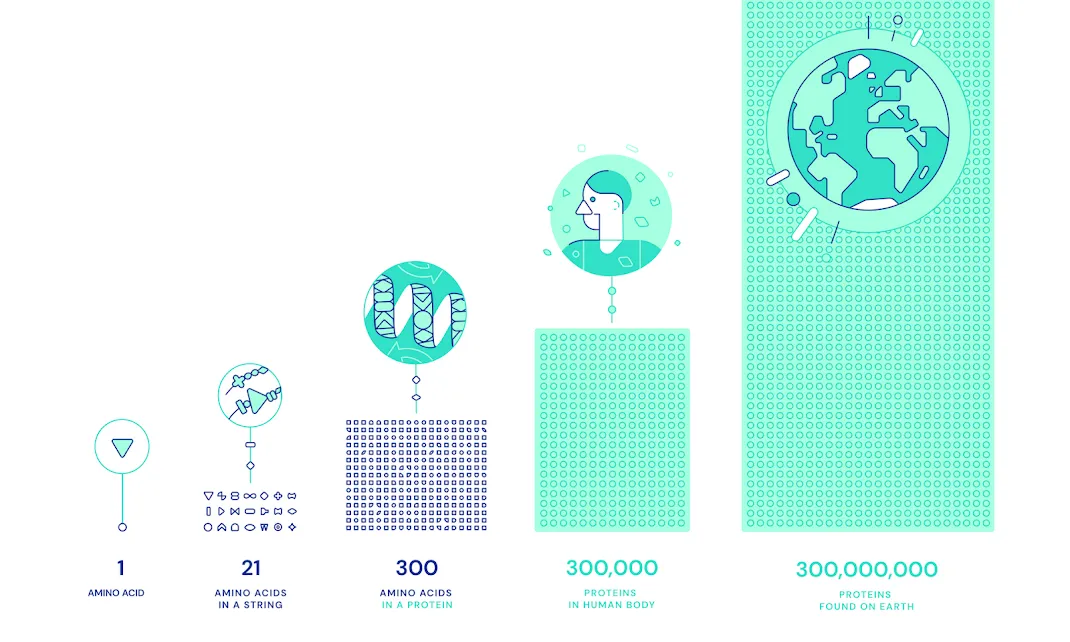
蛋白质是由氨基酸序列组成的,人体内有21种氨基酸种类,但这21种氨基酸能够构成千万种蛋白质,但目前已知结构的蛋白质才2万个。
在人体细胞中,细胞核内有一个带有遗传信息的分子叫DNA,DNA中包含构造蛋白质的指令。
RNA通过一个被称为转录的过程复制DNA信息,这个带有遗传信息的RNA被称为mRNA,mRNA穿过细胞核来到细胞质内。
细胞质内还一个叫做核糖体的东西,蛋白质就是在这里合成的。
mRNA首先来到核糖体这里,细胞质内还有另一种RNA,叫做tRNA,tRNA一端是碱基对,另一端是氨基酸,当tRNA的碱基对和mRNA的碱基配对后,就会留下氨基酸,氨基酸就会串联在一起,最后进行折叠,形成一个3D结构的蛋白质。
蛋白质合成是细胞分裂过程中的重要活动,细胞构成组织,组织构成器官。人体内的很多酶也是由蛋白质构成的,人体的眼睛为什么能感光?肝脏为什么能造血?这都与具有某种结构的蛋白质有关。
所以,了解了蛋白质的结构,也就知道了它的功能,进而进行疾病预防,药物研发,甚至发现遗传密码。
二、传统分析蛋白质折叠的方法
仅基于氨基酸序列预测蛋白质的3D结构难度是巨大的,约有10的143次方种折叠方式,这比宇宙中的原子个数还要多。
传统预测蛋白质结构的方法主要有以下几种:
- 同源建模(Homology modeling):也称为比较建模或模板基础建模,基于蛋白质的3D结构比它们的氨基酸序列更为保守的假设。这种方法主要使用序列比对和分子建模技术,通过已知结构的同源蛋白来预测目标蛋白的结构。
- 从头建模(De novo modeling):这是一种基于“第一原理”的蛋白质结构预测方法,不依赖于已知的蛋白质结构,而是仅基于物理定律(量子力学)生成目标蛋白的3D结构。从头建模方法通过设计的能量函数引导构象搜索,选择具有最低能量的构象。
- 基于机器学习(ML)的建模:这是一种利用ML算法和已知蛋白质结构来预测目标蛋白质结构的策略。尤其是深度学习(DL)在这一领域取得了快速发展,DL基于大量数据和强大的计算能力,通过优化算法来预测蛋白质结构。
数百年来,为了预测蛋白质结构,需要借助磁共振核,X线等昂贵设备,同时需要消耗大量的时间进行实验。
为了解决这个世纪难题,还成立了一个CASP挑战赛。
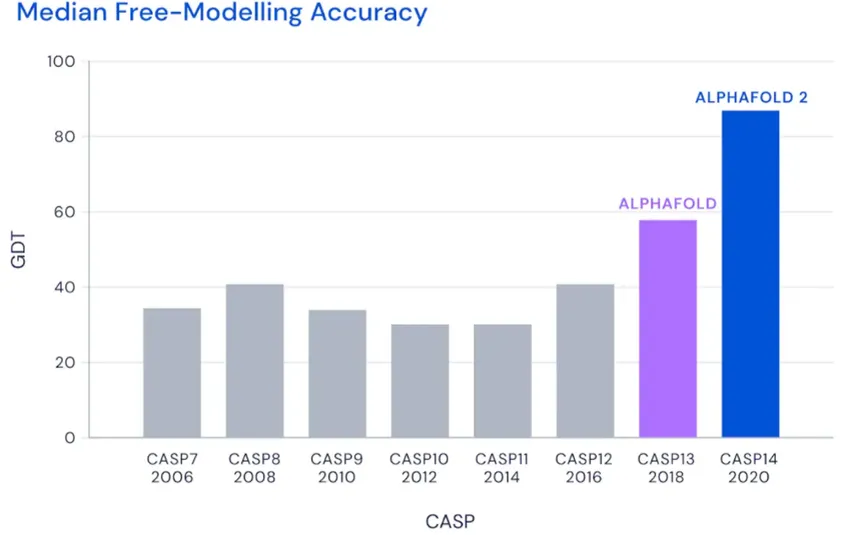
目前公认为,如果GDT达到85%就认为解决了蛋白质折叠世纪难题,如上图所示,ALPHAFOLD2的效果已经接近这个目标。而它的前任也远超传统方法。
三、Alphafold2
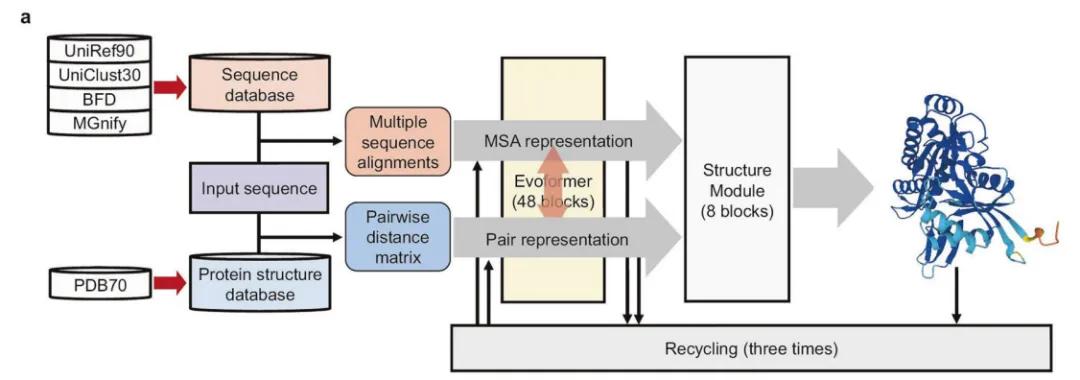
Alphafold2使用深度神经网络利用同源蛋白和多序列比对的信息从氨基酸序列生成蛋白质结构。
Github:https://github.com/google-deepmind/alphafold
论文:https://www.nature.com/articles/s41586-021-03819-2
Colab Notebook:https://colab.research.google.com/github/sokrypton/ColabFold/blob/main/AlphaFold2.ipynb模型结构
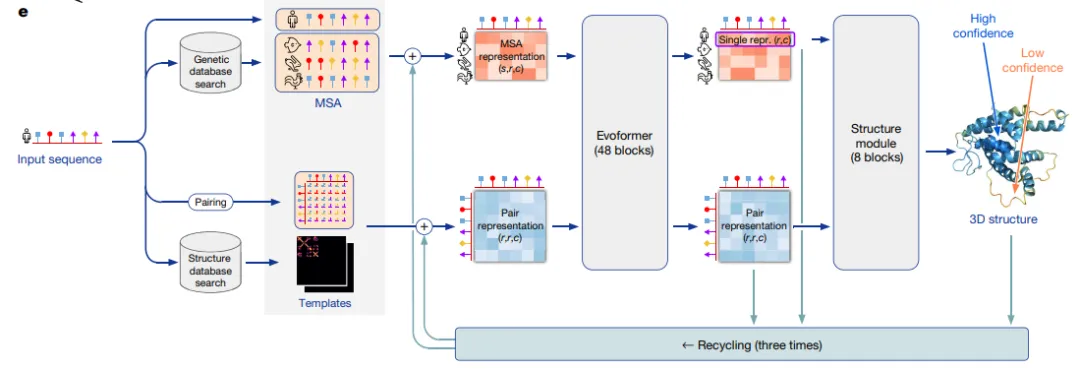
如下图,首先根据输入氨基酸序列和已知结构蛋白质数据库生成MSA(多序列对比表示)和配对表示。
MSA表示和配对表示会被送入Evoformer,它实现了从输入信息到蛋白质的3D原子坐标的转换。
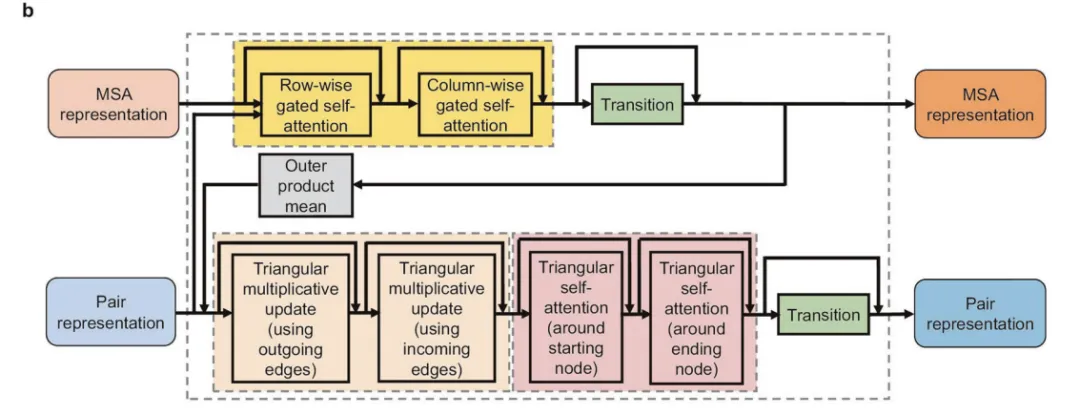
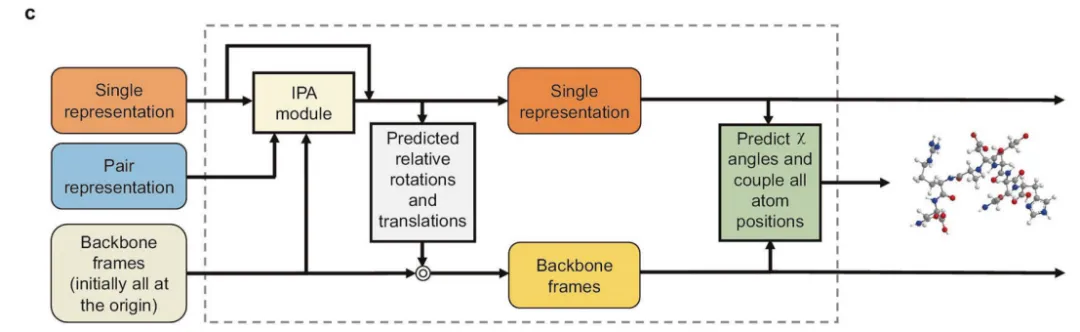
对于Alphafold2详细信息,感兴趣的可以去阅读论文。
这里只是简单概述一下,模型的输入是组成蛋白质的氨基酸序列,输出是PDB格式的数据,用于表示蛋白质3D结构信息。模型则是采用当下最流行的Transformer架构。
MSA是将输入蛋白质氨基酸序列与已知结构的蛋白质进行对比,目的是找到与之相似的,然后基于这个相似的蛋白质结构来预测输入蛋白质结构。
为了造福人类,DeepMind将一些蛋白质的预测结果收录数据库供其他组织或个人免费使用。
https://alphafold.ebi.ac.uk
本文转载自公众号人工智能大讲堂

















