CVPR 2024 Oral:生命之树大模型 原创
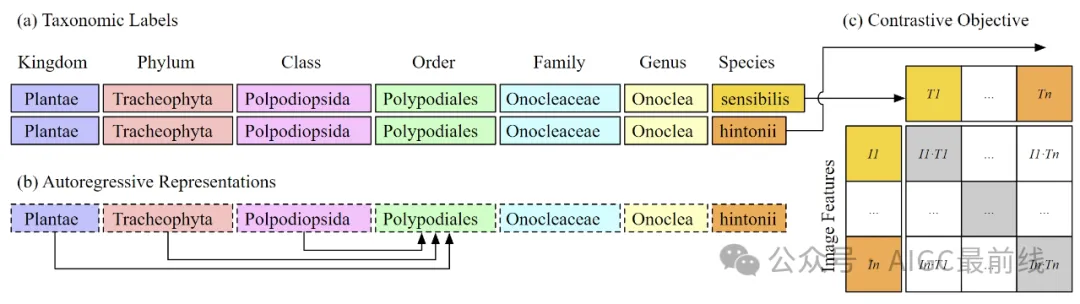
图1:使用CLIP目标(c)来训练一个ViT-B/16模型,该模型使用超过45万个不同的类标签,所有这些标签都来自生命之树的分类标签(a)。由于文本编码器是一个自回归语言模型,因此排序表示仅能依赖于高级别的分类,如类、门和界(b)。这自然导致了标签的层次表示,有助于视觉编码器学习更符合生命之树的图像表示。
自然界的图像是生物信息的超丰富来源。有许多计算方法和工具,特别是计算机视觉,可以从图像中提取信息。但是,现有的方法是针对特定任务的定制模型,并且不能适应或扩展到新的问题、情境和数据集。
近日,研究人员开发了第一个大规模多模态模型,BioCLIP,用于图像上的一般生物学问题。利用生物学作为计算机视觉应用领域的独特特性:
- 植物、动物和真菌图像的丰富性和多样性;
- 丰富的结构化生物知识的可用性。
该工作入选CVPR 2024 Oral。作者发布了一个大规模、多样化的生物图像数据集:TreeOfLife-10M,训练了BioCLIP,并在多样化的细粒度生物分类任务上对该方法进行了严格的基准测试,发现BioCLIP始终显著优于现有基线,绝对值提高了17%到20%。内在评估进一步揭示了BioCLIP已经学习到符合生命之树的层次化表示,从而突显了其强大的泛化能力。
实验
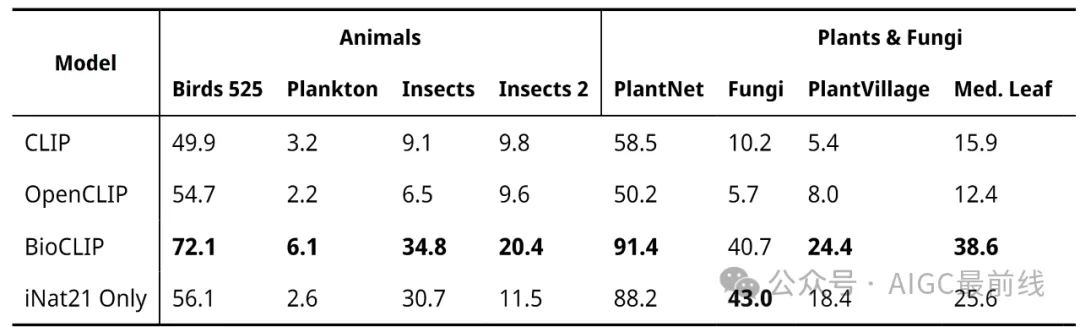
在各种各样的与生物学相关的分类任务上,作者评估了BioCLIP和三个基准模型,包括CLIP、OpenCLIP,以及仅使用与BioCLIP相同过程但仅在iNat21上训练的iNat-only模型。作者对所有模型进行了零样本分类,并在验证集上报告准确率。加粗表示每个任务的最佳性能。
实验结果表明,BioCLIP优于通用领域的基准模型以及iNat-only模型。
内在评估
为什么BioCLIP效果这么好?作者进行了内在评估,以了解BioCLIP学习到的表示。使用T-SNE在二维平面上绘制了iNat21验证集中10万个未见过的图像的BioCLIP和CLIP的表示,并根据它们的类别对点进行着色。在下图中,(B)表示BioCLIP,(O)表示OpenAI的CLIP。
在类群等较高级别上,CLIP和BioCLIP都有良好的分离性,但您可以看到BioCLIP的表示更加细粒化,并包含更丰富的聚类结构。在较低级别上,BioCLIP产生了更具可分辨性的特征,而CLIP的特征往往混乱且缺乏清晰的结构。这表明BioCLIP已经学习到了符合分类学层次结构的丰富特征表示,这有助于解释其在生命之树上强大的泛化能力。
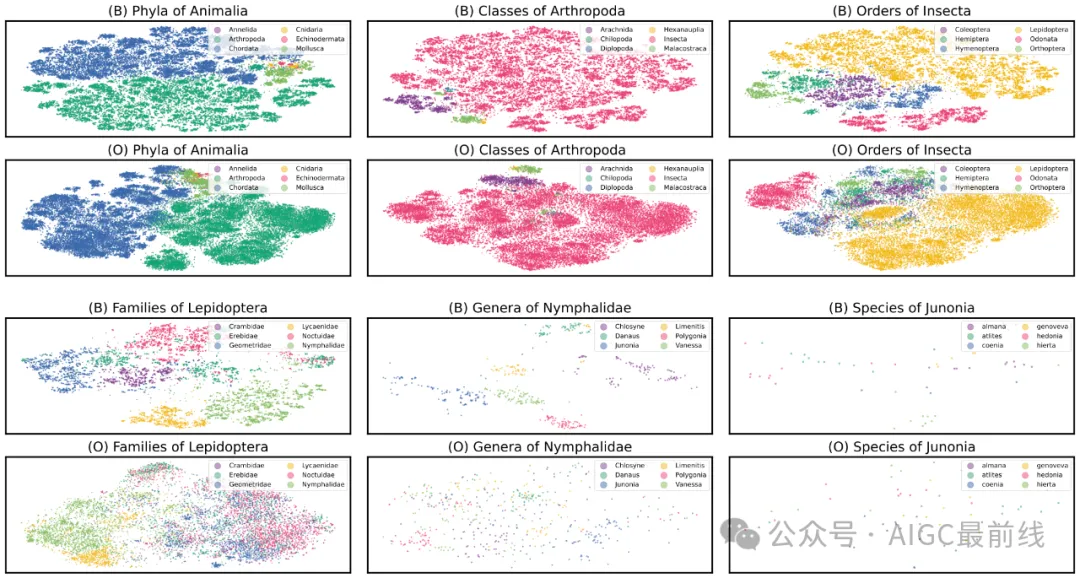
BioCLIP的表示比OpenAI的CLIP更细粒化,并且包含更丰富的聚类结构。
数据集

TreeOfLife-10M是目前可用的最大和最多样化的生物图像数据集。研究人员从三个来源,包括iNaturalist、BIOSCAN-1M和Encyclopedia of Life中获取图像,创建了一个包含1000万张图像的数据集,涵盖了45万多个物种。
参考:
@article{stevens2023bioclip, title={BioCLIP: A Vision Foundation Model for the Tree of Life}, author={Samuel Stevens and Jiaman Wu and Matthew J Thompson and Elizabeth G Campolongo and Chan Hee Song and David Edward Carlyn and Li Dong and Wasila M Dahdul and Charles Stewart and Tanya Berger-Wolf and Wei-Lun Chao and Yu Su}, year={2023}, eprint={2311.18803}, archivePrefix={arXiv}, primaryClass={cs.CV} }
本文转载自公众号AIGC最前线
原文链接:https://mp.weixin.qq.com/s/KgvscZ321hvB-oN2VB7yDQ
















