使用语言模型模拟五亿年的进化历程 精华

一、结论写在前面
论文标题:Simulating 500 million years of evolution with a language model
论文链接:https://www.biorxiv.org/content/10.1101/2024.07.01.600583v1.full.pdf
超过三十亿年的进化历程将生物学图像编码到天然蛋白质的空间中。论文在由进化生成的token上训练的语言模型可以作为进化模拟器,生成与已知蛋白质相距甚远的功能性蛋白质。
论文提出ESM3,一种前沿的多模态生成语言模型,该模型能够推理蛋白质的序列、结构和功能。ESM3能够遵循复杂的多模态提示,并对生物学对齐高度响应。论文引导ESM3生成荧光蛋白,并采用了一种思维链方法。
在论文合成的生成物中,发现了一种亮度极高的荧光蛋白,其与已知荧光蛋白的相似度仅为58 q_o,而如此远距离的天然荧光蛋白之间的进化间隔超过五亿年。
二、论文的简单介绍
2.1 论文的背景
现存的蛋白质在数十亿年的自然进化过程中演化成现今的形态,历经了漫长的进化筛选。在地质时间尺度上进行的平行实验中,自然界创造随机突变并应用选择,通过其多样的序列、结构和功能筛选蛋白质。
因此,论文在蛋白质中观察到的模式反映了那些深藏不露的生物学隐变量,这些变量随着时间的推移塑造了它们的演化。地球自然多样性的基因测序调查正在编录蛋白质的序列和结构,包含数十亿条序列和数亿个结构,揭示了生命间的变异模式。目前形成了一种共识,即在这些序列之下存在着一种基本的蛋白质生物学语言,可以利用语言模型来理解。
现已开发并评估了多种蛋白质序列的语言模型。研究发现,语言模型中涌现的表征反映了蛋白质的生物结构和功能,并且这些表征是在无监督情况下学习得到的,与这些属性无关,随着规模的扩大而改进。在人工智能领域,发现了预测能力随规模增长的缩放定律,描述了计算、参数和数据的前沿。
论文介绍了ESM3,一种前沿的多模态生成模型,该模型能够推理蛋白质的序列、结构和功能。ESM3被训练为针对每种模态的离散token的生成式掩码语言模型。通过将三维原子结构编码为离散token,而不是采用近期预测性和生成性模型中使用的复杂架构和三维空间扩散,实现了结构推理。对离散token的全对全建模是可扩展的,并允许ESM3根据其模态的任意组合进行提示,从而实现对新蛋白质的可控生成,这些新蛋白质能够尊重提示的组合。
ESM3在其最大规模上,通过1.07 × 10^24次浮点运算(FLOPs)对27.8亿个蛋白质和7710亿个独特token进行了训练,并拥有980亿个参数。将ESM3扩展到980亿参数规模,不仅提升了序列、结构和功能的表示能力,还在生成评估方面取得了改进。论文观察到ESM3对提示极为敏感,并能创造性地解决复杂提示组合的问题,包括那些在自然界中找不到匹配结构的问题。所有规模的模型都可以通过调整更好地遵循提示,而较大的模型在调整后对提示的响应更为显著,显示出解决最困难提示的更大能力。
论文报告了使用ESM3生成的一种新型绿色荧光蛋白(GFP)。荧光蛋白负责水母和珊瑚的发光颜色(29),是现代生物技术中的重要工具(30)。它们共享一个优雅的结构:一个由十一股β桶和一个穿过中心的螺旋组成的结构,该结构作为支架,促使蛋白质自身原子形成发光的发色团。这种机制在自然界中是独一无二的——没有其他蛋白质能自发地从其自身结构中形成荧光发色团——这表明即使对于自然界而言,产生荧光也是一项艰巨的挑战。
论文的新蛋白质,命名为esmGFP,与水母绿色荧光蛋白(Aequorea victoria GFP)的序列相似度为36%。尽管绿色荧光蛋白作为蛋白质工程的目标已受到数十年的广泛关注,据论文所知,如此远距离的新型GFP仅通过自然界中的发现获得。
在可预测的时间尺度上,自然界中的GFP经历了类似的多样化过程。从这个角度理解,在现有蛋白质如此远距离生成新的荧光蛋白似乎等同于模拟超过5亿年的进化历程。
2.2 ESM3
ESM3 模型在蛋白质的序列、结构和功能上进行推理。这三种模态均由token表示,并以独立轨道形式输入和输出,在模型内部融合成单一的潜在空间。ESM3 采用生成式掩码语言建模目标进行训练:
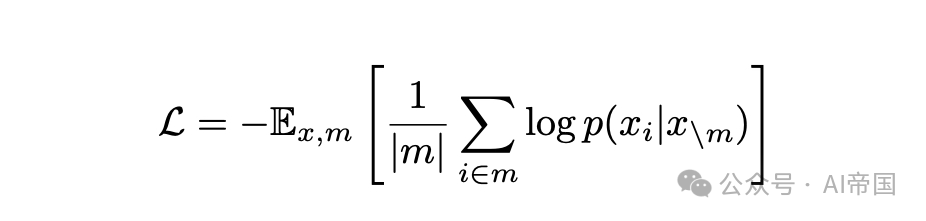
对描述蛋白质的token x 应用随机掩码 m,模型被监督以预测被掩码的token身份。在训练过程中,掩码采用噪声调度采样,变化掩码位置的比例,使得ESM3能够看到多种不同的掩码序列、结构和功能组合,并从任何其他模态预测任何模态的完成。这与传统的掩码语言建模不同,后者在所有可能的掩码率上应用监督,而不是单一固定掩码率。这种监督机制分解了给定任意先前token组合下所有可能的下一个token预测的概率分布,确保了可以从任意起点以任意顺序生成token。
为了从ESM3生成token,token是迭代采样的。从完全或部分掩码的上下文开始,token可以在任何顺序下逐个或并行采样,直到所有位置完全未掩码。除了实现生成功能外,ESM3的训练目标也适用于表示学习。较高的掩码率提高了生成能力,而较低的掩码率则提高了表示学习的效果。论文选择使用一种平衡生成能力和表示学习的噪声调度来训练ESM3。
ESM3是一个双向Transformer模型。序列、结构和功能token在输入时嵌入并融合,然后通过一系列Transformer块进行处理(图1B)。在模型的输出端,浅层多层感知器(MLP)头将最终层表示投影为每个轨道token的概率。ESM3通过token化,而不是专门的架构组件,来表示蛋白质在学习的多元特征空间中的复杂性。这使得训练既高效又高度可扩展。
蛋白质结构通过离散自编码器进行token化处理,该编码器训练用于将三维结构压缩为离散token(图1C)。论文提出了一种不变几何注意力机制,以高效处理三维结构。该机制在每个氨基酸的键几何结构定义的局部参考框架内运行,并通过转换为全局框架,允许局部框架进行全局交互。围绕每个氨基酸的局部结构邻域被编码为一系列离散token,每个氨基酸对应一个token。
在预测或生成蛋白质结构时,ESM3输出的结构token通过解码器传递,解码器重建完整的原子结构。自编码器训练用于编码和重建原子坐标,采用几何损失函数监督键向量和法线的成对距离及相对方向。
由于每个结构token的局部邻域包含有关结构邻近部分的信息,论文还提供了一种机制,使模型能够通过第一个变换器块中的几何注意力直接基于主链原子坐标进行条件化。为了支持结构的高级抽象,论文包含了二级结构(SS8)token和溶剂可及表面面积token的轨道。描述生物学功能的关键词token,如结合、酶功能以及域或折叠分类,允许对蛋白质结构和功能进行更高层次的语义描述。残基水平注释token指示单个残基的功能,如催化位点和翻译后修饰。
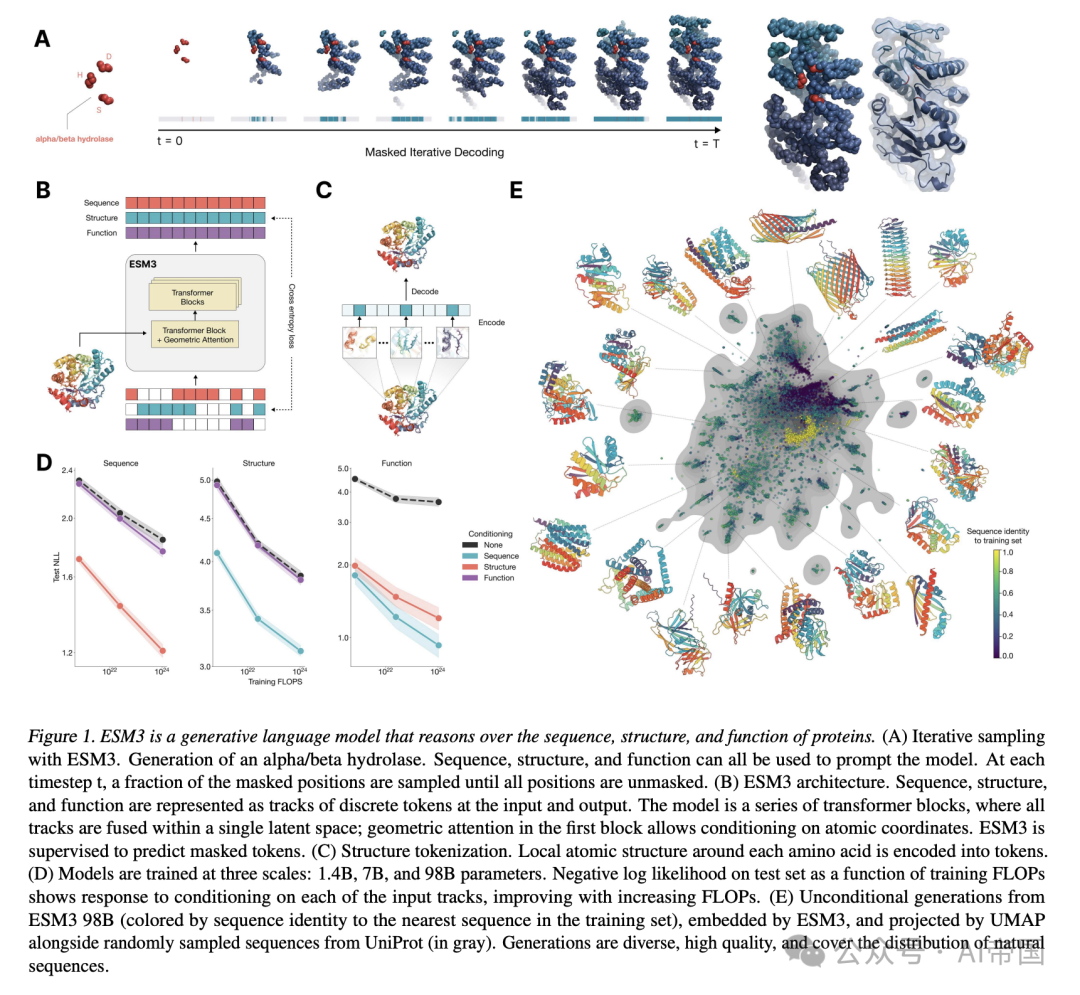
图1。LSM3 是一种生成式语言模型,能够推理蛋白质的序列、结构和功能。(A) 使用ESM3进行迭代采样。生成一个α/β水解酶。序列、结构和功能均可作为模型的提示。在每个时间步 t,一部分被遮蔽的位置会被采样,直到所有位置都未被遮蔽。(B) ESM3架构。序列、结构和功能在输入和输出中表示为离散token的轨迹。模型由一系列变换器块组成,所有轨迹在一个单一的潜在空间中融合;第一个块中的几何注意力允许基于原子坐标进行条件化。ESM3受到监督,以预测被遮蔽的token。(C) 结构token化。围绕每个氨基酸的局部原子结构被编码成token。(D) 模型在三个规模上进行训练:1.4亿、7亿和98亿参数。测试集上的负对数似然作为训练浮点运算次数的函数,显示了模型对每个输入轨迹条件化的响应,随着浮点运算次数的增加而改进。(E) 来自ESM3 98B的无条件生成序列(根据与训练集中最近序列的序列同一性着色),通过ESM3嵌入,并由UMAP投影,同时随机从UniProt中采样的序列(以灰色表示)。生成序列多样性高、质量好,并且覆盖了自然序列的分布
最大的ESM3模型是在从序列和结构数据库中收集的27.8亿个自然蛋白质上进行训练的。由于相对于序列,只有一小部分结构是通过实验确定的,论文利用了预测的结构。序列通过使用隐马尔可夫模型库(40)注释了功能关键词。论文还为所有结构(包括预测的结构)生成合成序列,使用了一个逆折叠模型。总体上,这使得训练数据增加到31.5亿个蛋白质序列,2.36亿个蛋白质结构,以及5.39亿个带有功能注释的蛋白质,总计7710亿个独特token。
论文对ESM3模型进行了三种规模的训练:14亿、70亿和980亿参数。在一系列初步实验中,为了评估架构超参数对表征学习性能的影响,论文发现增加深度比增加宽度有更大的响应。这促使论文选择了相对较深的网络作为最终架构,其中980亿参数模型包含了216个Transformer块。
将ESM3从1.4亿参数扩展到980亿参数,在测试集的所有轨迹上均带来了显著的损失改进,其中序列损失的改进最为显著(图1D)。这些测试损失的提升导致了更好的表征学习(表S7和图S8)。在单序列结构预测中,ESM3 98B超越了ESMFold(0.895 vs. 0.865平均局部距离差异测试,LDDT,CAMEO测试集;)。从模型中无提示生成(无条件生成)的序列产生了高质量的蛋白质——平均预测LDDT(pLDDT)为0.84,预测模板建模得分(pTM)为0.52——这些蛋白质在序列(平均成对序列同一性0.155)和结构(平均成对TM得分0.48)上均具有多样性,涵盖了已知蛋白质的分布范围(图1E,图S13)。
尽管已有大量研究致力于为蛋白质创建专门的架构和训练目标,论文的结果表明,通过token化、高效架构和掩码token预测实现的基于语言模型的扩展,在表征和生成应用中持续带来了改进。这种方法使得模型能够构建一个从数据中学习的多模态共享表征空间,而非显式地硬编码到其架构中,这使得在计算和数据不断增加的情况下,模型能够学习到一个越来越丰富和通用的特征空间。在接下来的章节中,论文将展示这种方法在蛋白质的可控生成中实现了高保真度。
2.3 可编程设计与ESM3
论文探究了ESM3遵循不同组合复杂提示的能力。ESM3可以接收来自其各个输入轨迹的指令提示:序列、结构坐标、二级结构(SS8)、可溶性表面积(SASA)以及功能关键词。这使得提示可以在多个抽象层次上指定,从原子级别的结构到描述功能和折叠拓扑的高级关键词。
论文独立评估ESM3在每个赛道中遵循提示的能力(图2A)。为每个赛道构建一组提示,使用从自然蛋白质中暂时保留的测试集。生成的结果使用ESMFold进行评估,以确保与提示的一致性和结构预测的置信度(pTM)。论文为每个赛道定义了一致性指标:受限位点RMSD(cRMSD),即提示坐标(即主链原子的位置)与生成中相应坐标之间的RMSD;SS3准确性,提示与生成之间三类二级结构匹配的残基比例;SASA ρ,SASA提示与生成相应区域之间的相关性;以及关键字恢复,InterProScan恢复的提示关键字比例(40)。在所有赛道中,7B参数的ESM3找到了遵循提示且结构被ESMFold自信预测的解决方案(pTM > 0.8)。
无条件生成反映了自然蛋白质的分布。由于论文观察到ESM3能够忠实地遵循提示,论文推测提示可以引导模型生成与训练集和自然蛋白质不同的蛋白质。首先,论文测试模型遵循分布外提示的能力。论文构建了一组结合SS8和SASA的提示,来自保留结构(TM < 0.7至训练集)。在这些提示下,尽管模型继续生成连贯的球状结构(平均pTM 0.85 ± 0.03),但与训练集相似度的分布(以TM-score和序列同一性衡量)变得更加新颖(与最近训练集蛋白质的平均序列同一性<20%,平均TM-score 0.48 ± 0.09)。为了测试模型对超出自然蛋白质分布结构的泛化能力,论文使用从人工对称蛋白质设计数据集中提取的二级结构提示,这些设计与训练数据集中的自然蛋白质不同。同样,ESM3生成了高置信度的生成(pTM > 0.8,pLDDT > 0.8),与训练集中的蛋白质在序列和结构上相似度低(序列同一性<20%,TM-score 0.52± 0.10),表明模型可以用于生成与自然界存在高度不同的蛋白质序列和结构。
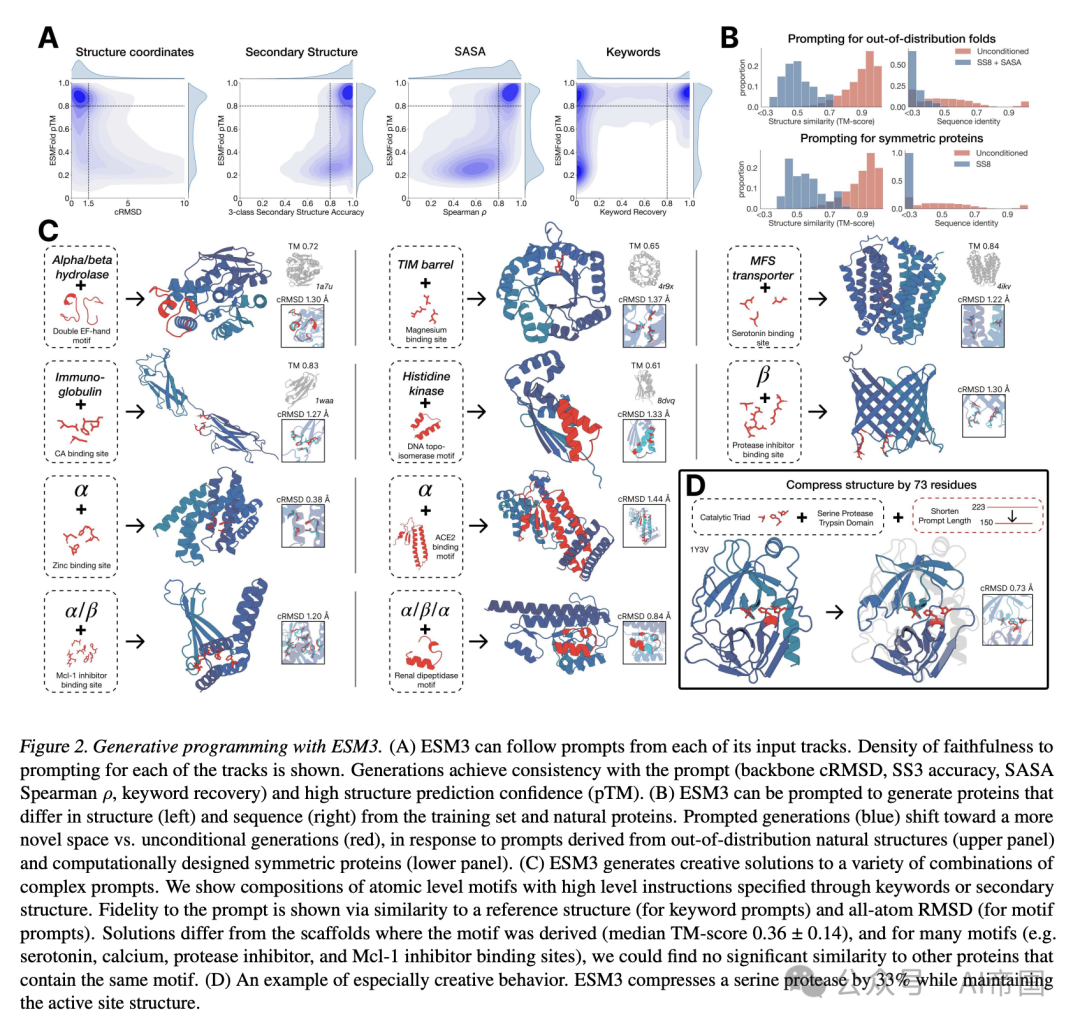
图2. 使用ESM3进行生成式编程。(A) ESM3能够遵循其输入轨道的提示。每个轨道的提示忠诚度密度如图所示。生成结果与提示保持一致(骨架cRMSD、SS3准确性、SASA Spearman ρ、关键词恢复),并具有高结构预测置信度(pTM)。(B) ESM3能够根据提示生成在结构(左)和序列(右)上与训练集和自然蛋白质不同的蛋白质。有提示的生成(蓝色)相对于无提示的生成(红色),在响应来自分布外自然结构(上部面板)和计算设计的对称蛋白质(下部面板)的提示时,向更新的空间转移。(C) ESM3能够为各种复杂提示的组合生成创造性解决方案。论文展示了通过关键词或二级结构指定的高级指令与原子级 motifs 的组合。提示的忠诚度通过与参考结构的相似性(关键词提示)和全原子RMSD(motif提示)来展示。解决方案与从中提取motif的支架不同(中位TM-score 0.36± 0.14),对于许多motifs(例如血清素、钙、蛋白酶抑制剂和Mcl-1抑制剂结合位点),论文找不到与包含相同motif的其他蛋白质有显著相似性。(D) 一个特别具有创造性的行为的例子。ESM3将一种丝氨酸蛋白酶压缩了33%,同时保持了活性位点结构
ESM3能够遵循复杂的提示,并有能力从不同轨道和不同抽象层次组合提示。为了评估这一能力,论文向ESM3提供需要解决单个原子空间协调的motifs,包括参与序列中相距较远的残基之间的三级接触的原子,如催化中心和配体结合位点。论文将motif提示与指定折叠架构的提示相结合。对于每个独特的motif和支架组合,论文生成样本,直到成功(全原子RMSD < 1.5 AA,包括侧链原子的原子坐标;TM > 0.6 到折叠级提示的代表性结构;SS3 准确性 > 80% 对于二级结构提示;以及对支架的高置信度,pTM > 0.8,pLDDT > 0.8)。
论文发现ESM3能够解决多种此类任务(图2C)。它在不恢复原始支架的情况下完成这一任务(与参考蛋白的中位TM-score为0.40± 0.109)。在某些情况下,支架来自于具有相似基序的现有蛋白质(例如,为锌结合基序设计的ESM3-设计α螺旋支架与Ni_2+结合蛋白高度相似,PDB: 5DQW, 5DQY;图2C,行3列1)。对于许多基序(例如,血清素、钙、蛋白酶抑制剂和Mcl-1抑制剂的结合位点),Foldseek未发现与其他含有相同基序的蛋白质有显著相似性。在这些情况下,论文观察到有时基序被嫁接到完全不同的折叠结构中(例如,β桶中的蛋白酶抑制剂结合位点基序与膜结合铜转运蛋白最为相似,PDB: 7PGE;图2C,行3列3)。其他时候,支架似乎是完全新颖的,例如为Mcl-1抑制剂结合基序设计的α/β蛋白,其结构与PDB、ESMAtlas和AlphaFold数据库中的所有已知蛋白质的相似性都很低(最大TM-score < 0.5;图2C,行4列1)。总体而言,生成的解决方案具有高设计性,即在使用ESM-IF1(42)进行逆折叠和使用ESMFold进行重折叠后,能够自信地恢复原始结构(中位pTM 0.80± 0.08;scTM 0.96 - 0.04)。
通过实验性的提示工程,论文观察到对提示特别有创意的响应。在这里,论文重点介绍一个蛋白质压缩的例子(图2D)。从天然的胰蛋白酶(PDB 1Y3V)开始,论文使用催化三联体的序列和坐标以及描述胰蛋白酶的功能关键词进行提示,但将总体生成长度减少了三分之一(从223个残基减少到150个残基)。ESM3保持了活性部位的协调性(全原子RMSD 0.73A)和整体折叠的高设计性(pTM 0.84,scTM均值0.97,标准差0.006),尽管序列长度显著减少且折叠仅由功能关键词提示指定。
这些例子展示了ESM3在单独或组合使用其输入轨道中的任何提示时,找到创意解决方案的能力。这种能力使得蛋白质设计能够采用理性的方法,通过生成模型在提示和生物复杂性之间架起桥梁,从而在从高级拓扑结构到原子坐标的各个抽象层次上提供控制。
2.4 生物学对齐
虽然论文观察到基础模型性能随着规模的增长而有意义的提升,但更大的模型可能具有论文尚未观察到的更大潜在能力。基础ESM3模型可以被提示执行诸如三级 motif 支架搭建和提示组合等困难任务,尽管这些模型并未针对这些目标进行明确优化。由于论文评估生成输出的属性——如对提示的遵守程度或支架的置信度——仅在预训练期间间接被模型看到,通过微调将模型直接对齐到生成任务可能会在更大模型中引发更大的能力差异。
论文研究了如何将基础模型对齐(43, 44)以生成满足挑战性提示的蛋白质。对于每个模型,论文构建了一个包含连续残基片段和三级结构基序(也指定了接触氨基酸的身份)的主链原子坐标提示数据集。论文为每个提示生成多个蛋白质序列,并使用ESM3对每个序列进行折叠,评分依据与提示的一致性(主链cRMSD)和结构预测的置信度(pTM)。高质量样本与低质量样本配对,以构建偏好数据集。随后,ESM3通过偏好优化损失(45, 46)进行微调,使得模型相对于低质量样本更倾向于高质量样本。
在调整每个基础模型后,论文评估它们的绝对性能,以及生成分布的偏移。论文关注一系列需要协调三级接触中残基主干原子的具有挑战性的提示。论文评估生成高质量支架(pTM > 0.8)的能力,这些支架能够以高分辨率(主干cRMSD < 1.5A)遵循提示,使用ESM-Fold进行评估。论文使用从46个配体结合基序的保留数据集中提取的氨基酸身份和主干原子坐标来提示每个模型。对于每个基序,论文通过排列残基顺序、改变其在序列中的位置以及改变序列长度,创建了1024个提示。每个提示生成一个蛋白质。每个基序的1024个生成结果用于构建一个无偏估计量,估计在128次生成后解决三级协调任务的比例。
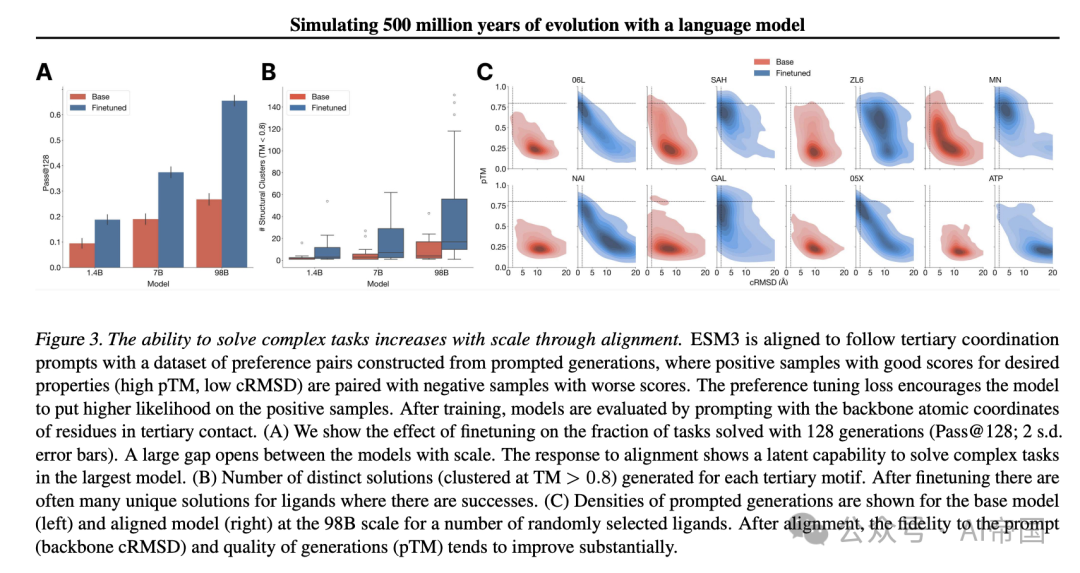
图3. 通过对齐,解决复杂任务的能力随着规模增加而增强。ESM3通过一个由提示生成构建的偏好对数据集来遵循三级协调提示,其中具有良好得分(高pTM,低cRMSD)的正样本与得分较差的负样本配对。偏好调谐损失鼓励模型对正样本赋予更高的似然。训练后,模型通过提示包含三级接触中残基的骨架原子坐标来进行评估。(A)论文展示了在128次生成中解决任务的比例(Pass@128;2个标准差误差棒)的微调效果。随着模型规模的扩大,两者之间出现了显著差距。对齐响应显示,最大模型中潜藏着解决复杂任务的能力。(B)为每个三级基序生成的独特解决方案数量(在TM > 0.8 处聚类)。微调后,对于成功的配体,通常存在许多独特的解决方案。(C)对于多个随机选择的配体,基础模型(左)和对齐模型(右)在98B规模上的提示生成密度显示。对齐后,生成结果对提示的忠实度(骨架cRMSD)和质量(pTM)往往有显著提升。
对齐模型解决的协调任务数量是基础模型的两倍(图3A)。尽管基础模型在解决任务的百分比上存在差异(1.4B为9.5%,7 B为19.0%,98B为26.8%;图3A),但通过对齐揭示的能力差异更为显著(分别从9.5%增加到18.8%,19.0%增加到37.49%,以及26.8%增加到65.5%)。偏好调优模型不仅解决了更大比例的任务,而且在每个任务中找到了更多的解决方案,这是通过不同结构簇的数量来评估的(TM > 0.8,主链cRMSD < 1.5 AA,且pTM > 0.8;图3B)。观察到ESMFold pTM和主链cRMSD在每个配体结合基序的分布上发生了变化(图3C;图S17)。在98B规模上,微调模型在37个测试配体中的46个上产生了比基础模型更多的独特成功簇,而剩下的9个配体未被基础模型或对齐模型解决,这表明对齐几乎普遍提高了生成蛋白质的结构预测的忠实度和置信度。与仅最大化正样本似然的监督微调基线相比,偏好调优在所有规模上都带来了更大的改进。
论文的实验揭示了模型规模之间在能力上存在显著差异。最大的对齐模型相对于对齐前的基础模型以及对齐后的小模型都有了显著的改进。通过对齐,模型学会了从少量示例中进行泛化:生成的分布发生了变化,以提高支架的质量和与提示的一致性,增加了解决任务的比例和不同解决方案的数量。
对齐要求模型通过示例进行学习。模型能够识别微调示例所展示的底层属性,并将这些演示泛化到新任务中,这意味着微调能够访问这些属性的内部表示。这个表示空间是通过预训练过程学习的,其中模型在跨越进化的蛋白质上进行训练,这表明它反映了并包含了蛋白质生物学的巨大多样性和复杂性。这样的表示空间很可能包含支持多种生物属性泛化的特征。较大模型对对齐的更高响应性表明,它们的内部表示空间更好地近似于那些底层属性,这是通过预训练中学习的特征实现深度迁移能力的证据,这种能力随着规模的增加而提高。
2.5 生成一种新的荧光蛋白
论文试图理解基础预训练的ESM3模型是否具有足够的生物学保真度来生成功能性蛋白质。论文着手创建一种与现有荧光蛋白序列相似度低的、具有功能性的绿色荧光蛋白(GFP)。论文选择荧光功能,因为它难以实现,易于测量,并且是大自然中最美丽的机制之一。
负责水母荧光和珊瑚鲜艳色彩的GFP家族蛋白,具有独特的生物学特性。
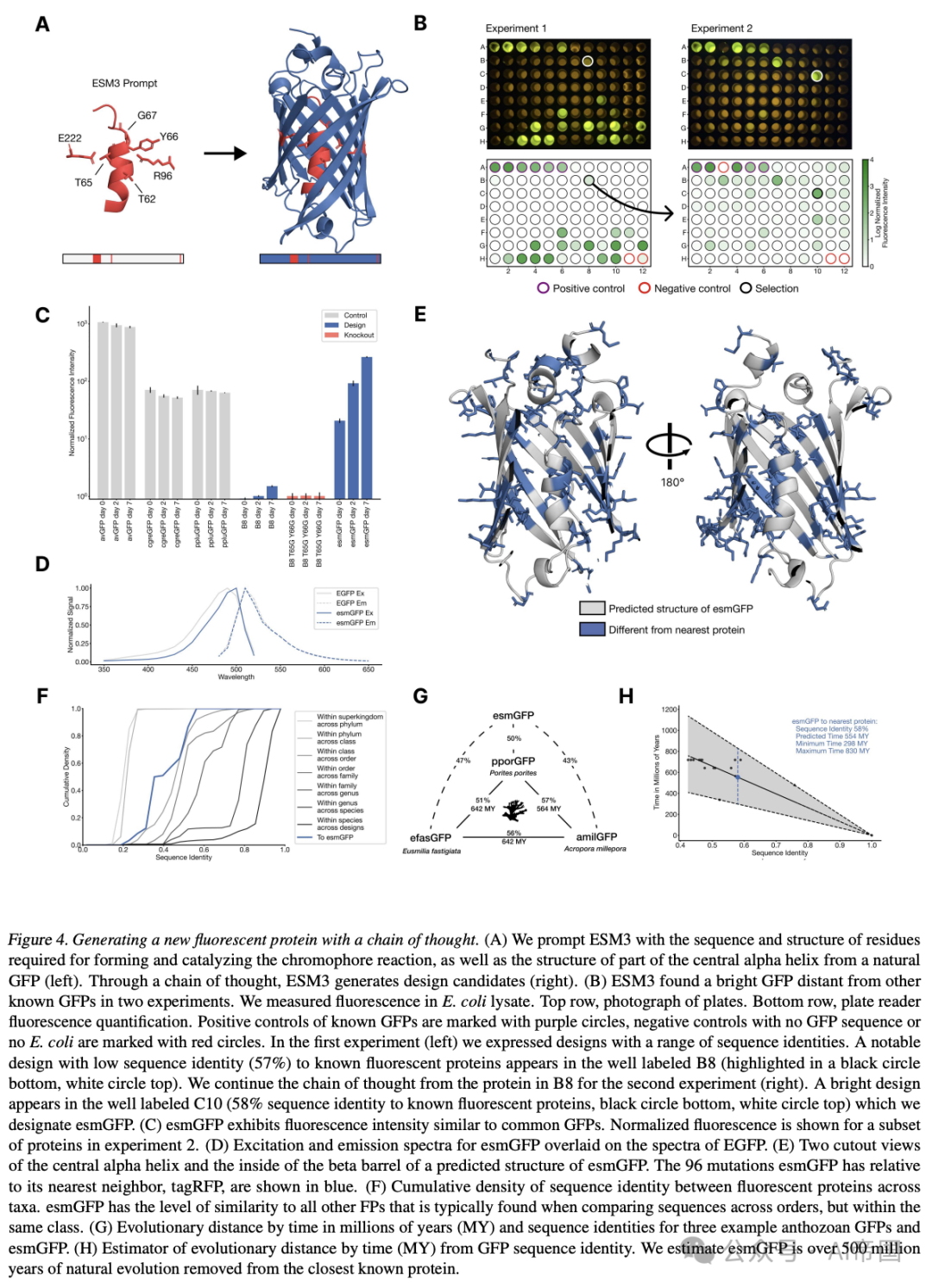
图4. 通过思维链生成新型荧光蛋白。(A) 论文向ESM3提供了形成和催化发色团反应所需的残基序列和结构,以及天然GFP中心α螺旋的一部分结构(左)。通过思维链,ESM3生成了设计候选方案(右)。(B) ESM3在两次实验中找到了一个在亮度上远离其他已知GFP的亮GFP。论文在E大肠杆菌裂解液中测量了荧光。顶部行,平板照片。底部行,平板阅读器荧光定量。已知GFP的阳性对照用紫色圆圈token,无GFP序列或无E大肠杆菌的阴性对照用红色圆圈token。在第一次实验(左)中,论文表达了具有一系列序列同一性的设计。一个与已知荧光蛋白序列同一性较低(57%)的显著设计出现在token为B8的孔中(底部黑圈,顶部白圈)。论文继续从B8中的蛋白质进行第二次实验(右)。一个亮设计出现在token为C10的孔中(与已知荧光蛋白序列同一性为58%,底部黑圈,顶部白圈),论文将其命名为esmGFP。(C) esmGFP表现出与常见GFP相似的荧光强度。实验2中部分蛋白质的归一化荧光显示。(D) esmGFP的激发和发射光谱与EGFP的光谱叠加。(E) esmGFP预测结构的中心α螺旋和β桶内部的两个切割视图。esmGFP相对于其最近邻tagRFP的96个突变以蓝色显示。(F) 跨分类群荧光蛋白序列同一性的累积密度。esmGFP与其他FP的相似度通常在比较不同目之间的序列时发现,但在同一类别内。(G) 三个示例珊瑚GFP和esmGFP的进化距离(以百万年为单位)和序列同一性。(H) 根据GFP序列同一性估计的进化距离(以百万年为单位)。论文估计esmGFP与最接近的已知蛋白质相距超过5亿年的自然进化
在无需辅因子或底物的情况下形成荧光发色团的能力(30)。这一特性使得GFP序列能够被插入到其他生物的基因组中,用于可视化token分子、细胞结构或过程,提供了一个在生物科学领域广泛应用的基础工具包。
GFP家族已成为数十年来蛋白质工程研究的重点,但功能变体的多样性绝大多数仍源自对自然界的探索。理性设计和突变技术已产生具有改进特性的GFP序列——如更高的亮度或稳定性,或不同颜色的变体——这些变体通常只包含少量突变(通常为5到15个,总共238个氨基酸编码序列)。在少数情况下,借助高通量实验和机器学习,科学家能够在保留荧光特性的同时引入多达40-50个突变(即80%序列同一性)(48-50)。
生成新的GFP需要实现其荧光特性背后的复杂生物化学和物理过程。所有GFP中,一个自催化过程在蛋白质核心的三种关键氨基酸之间形成发色团。GFP独特的结构,即一个弯曲的中央α螺旋被具有向内协调残基的十一股β桶包围,促成了这一反应。形成后,发色团不仅需要吸收光,还必须发射光以实现荧光。光发射对发色团周围的局部电子环境极为敏感。GFP的适应度景观反映了实现其功能所需的活性位点和周围三级相互作用的精确配置,因为少数随机突变就足以将荧光降低至零。
为了生成新的GFP序列,论文直接引导基础预训练的7B参数ESM3模型,以Thr62、Thr65、Tyr66、Gly67、Arg96、Glu222这些对形成和催化发色团反应至关重要的残基为条件,生成一个229残基的蛋白质(图4A)。此外,论文还以1 QY3实验结构中58至71残基的结构为条件,这些残基已知对发色团结构能的稳定性具有重要影响(53)。具体而言,输入包括序列token、结构token以及骨架原子坐标,生成过程从一个几乎完全被遮蔽的229残基token数组开始,除了用于条件的token位置。
论文采用以下思维链过程生成设计:模型首先生成结构token,有效创建蛋白质骨架。具有活性位点充分良好原子协调但与1QY3骨架整体结构有所差异的骨架通过筛选进入链式过程的下一步。论文将生成的结构添加到原始提示中,以新提示为条件生成序列。随后进行迭代联合优化,交替优化序列和结构。论文拒绝那些失去活性位点原子协调的思维链。论文从生成协议的迭代联合优化阶段的中间和最终点抽取数十万个候选GFP设计。论文根据与已知荧光蛋白的序列相似性对设计进行分类,并利用多种指标进行筛选和排序。
论文进行了一项初步实验,涉及96孔板上的88种设计,评估了每个序列相似性桶中的顶级代。每个生成的蛋白质都被合成,在E大肠杆菌中表达,并在485 nm激发波长下测量其荧光活性(图4B左侧)。论文测量了一些设计的亮度,这些设计与天然存在的GFP具有更高的序列同一性,其亮度与阳性对照相似。论文还识别了一个位于B8孔的设计(以黑色圆圈高亮显示),其与1QY3序列的序列同一性仅为36%,与最近的现有荧光蛋白tagRFP的序列同一性为57%。该设计的亮度比天然GFP低50倍,且其发色团成熟过程需要一周时间,而非不到一天,但它在新的一部分序列空间中表现出功能信号,据论文所知,这在自然界或通过蛋白质工程尚未发现。
论文继续从B8孔设计的序列出发,通过相同的迭代联合优化和排序程序,生成亮度提高的蛋白质。论文创建了第二块96孔板的设计,并使用相同的板读取器测定法发现,这一批次中的少数设计具有与自然界中发现的GFP相当的亮度。第二块板上位于C10孔的最佳设计(图4B右侧),论文将其命名为esmGFP。
论文发现esmGFP在天然GFP的亮度分布中表现出色。论文评估了在色团成熟第0天、第2天和第7天的荧光强度,并将这些测量结果绘制为esmGFP、B8的重复、B8的色团敲除变体,以及三种天然GFP(avGFP、cgreGFP、ppluGFP)的图表(图4C)。esmGFP比论文测量的已知GFP成熟时间更长,但在两天后达到相当的亮度。为了验证荧光是通过预期的Thr65和Tyr66介导的,论文展示了当这些残基被突变为甘氨酸时,B8和esmGFP变体失去了荧光活性(图S21)。
尽管两种蛋白质的发射峰值均为512 nm(图4D)。光谱形状表明,esmGFP的激发光谱的半峰全宽(FWHM)更窄(esmGFP为39 nm,而EGFP为56 nm),而它们的发射光谱的FWHM高度可比(分别为35 nm和39 nm)。总体而言,esmGFP表现出与已知GFP一致的光谱特性。
接下来,论文试图了解esmGFP与已知蛋白质的比较情况。通过非冗余蛋白质序列数据库的BLAST(54)搜索和ESM3训练集的MMseas(55)搜索,报告的相同最高命中是tagRFP,这也是B8的最近邻,具有58%的序列同一性,代表序列中共有96个突变。tagRFP是一种设计变体,而从自然界中与esmGFP最接近的野生型序列是eqFP578,一种红色荧光蛋白,与esmGFP在107个序列位置上有所不同(53%同一性)。esmGFP与tagRFP之间的序列差异在整个结构中都有发生(图4E),其中22个突变发生在蛋白质内部,由于发色团接近和高密度相互作用,该区域已知对突变高度敏感(56)。
对648种自然和设计GFP样荧光蛋白的序列比对分析显示,esmGFP与其他所有FPs的相似度水平通常在跨分类目比较序列时发现,但在同一分类纲内(图4F)。例如,esmGFP与其他FPs的差异程度类似于石珊瑚目(石珊瑚)和海葵目(海葵)这两个都属于海洋无脊椎动物中更大的纲——珊瑚纲的FPs之间的差异水平(图4G)。与esmGFP最接近的FPs来自珊瑚纲(珊瑚和海葵;平均序列同一性51.4%),但esmGFP也与水螅纲(水母)中的FPs共享一定程度的序列同一性,其中avGFP被发现(平均序列同一性33.4%)。
论文可以从进化生物学中获得启示,了解通过自然进化产生具有相似序列同一性的蛋白质所需的时间。在图4G中,论文将esmGFP与三种刺胞动物的GFP并列展示。论文利用对刺胞动物进行的时间校准系统发育分析(参考文献5),估计了这些物种之间进化时间的百万年前(MYA)至最近共同祖先的时间。使用包含六个刺胞动物GFP和物种的数据集,这些物种的最近共同祖先的MYA和GFP序列同一性准确,论文构建了一个简单的估计器,将FP之间的序列同一性与物种之间的进化时间(MY)相关联(图4H),以校准自然进化。基于此分析,论文估计esmGFP代表了超过5亿年的进化等效时间。
本文转载自 AI帝国,作者: 无影寺

















